说起基因组印记(Genomic imprinting),小编的记忆还停留在上学帮师兄查印记基因(imprinted genes)数据库的时候。那个时候其实根本不懂印记到底是什么,只记住了师兄在耳边的碎碎念“父本印记则母本表达父本甲基化,母本印记则父本表达母本甲基化”。虽然后来做印记方面的工作少了,但是提起基因组印记还是倍感亲切(也可能是对当年彻夜的查数据库的耿耿于怀)。
今年2月份发表在核酸研究上的一篇工作就采用了10X左右的Nanopore测序的数据进行基因印记的相关研究。作者采用两种方法(Basecall method基于Fastq数据,Signal method基于原始电信号)对Nanopore测序的read进行单倍型分配,并识别父母本等位差异的甲基化区域。这些等位差异的甲基化区域中有部分是已知的印记控制区,同时结果与RNA-seq等位特异表达和RRBS数据结果吻合。因此作者认为那些新的等位差异甲基化区域可能是新的印记控制区。
关键结论
当当当,详细解读来啦~~
文章:Using long-read sequencing to detect imprinted DNA methylation
发表日期:2019年2月
杂志:Nucleic Acids Research
影响因子:11.561
研究背景
基因印记作为一个经典的表观遗传现象,是指基因的表达具有亲本选择的现象,即只一个亲本的等位基因表达,另一个亲本的等位基因不表达或很少表达。印记基因在生长发育中。众多假设认为DNA甲基化是基因组印记的主要机制,即许多的基因印记是由不同亲本来源的等位基因调控区的差异甲基化造成的。
研究方法
- RRBS: B6 X Cast
- RNA-seq:4个B6 X Cast正反交子代,4个Cast X B6正反交子代
- Nanopore:B6 X Cast, Cast X B6
研究结果
-
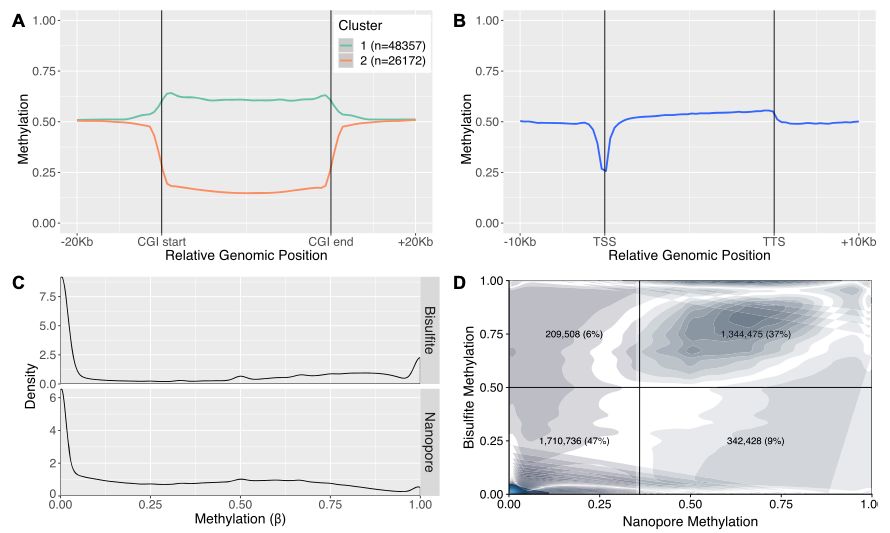
Nanopore甲基化结果与其他技术结果一致
对B6 X Cast杂交的F1子代的雄性胚胎(E14.5)采用MinION平台进行8X深度的测序,采用Nanopolish软件识别甲基化。转录起始位点处甲基化水平降低,且如先前报道一致,胎盘组织的全基因组甲基化水平在50%左右。采用RRBS对相同样品进行测序发现了两个技术平台的甲基化水平相似。
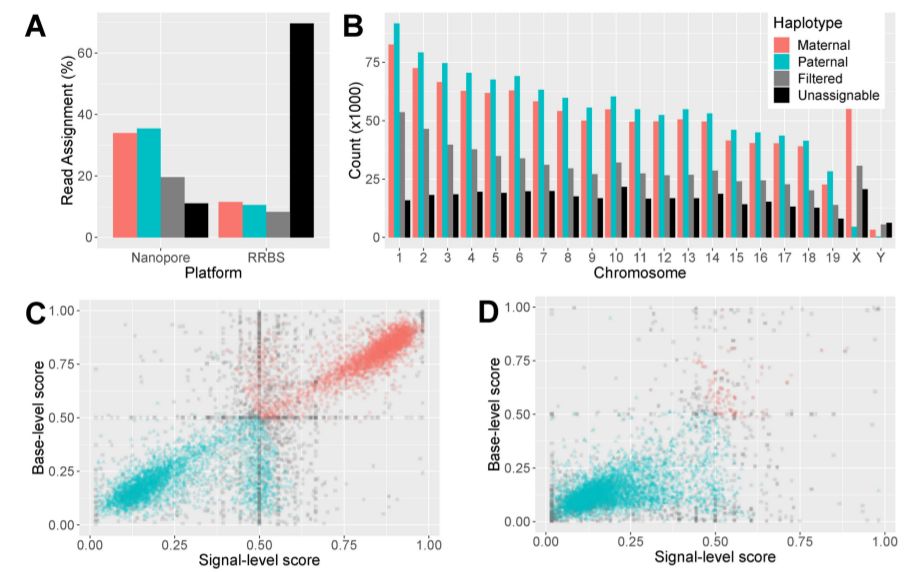
- Nanopore测序的read单倍型有效率更高
作者定义了两种分配单倍型的方法(Basecall method基于Fastq数据,Signal method基于原始电信号)。作者将测序数据与SNP标记的基因组进行比对,如果read上有至少5个高置信的SNP则将单倍型分配给Nanopore read,对于RRBS数据,只要有一个SNP即分配单倍型。结果发现只有24%的RRBS测序read被分配了单倍型,而74%的Nanopore测序的read可以被分配单倍型,且父母本等位各一半。同时对于雄性样品,比对到X染色体上绝大多数91%的read都被分配为母本单倍型。
为了进一步评估nanopore测序read分配单倍型的准确性,作者又测了B6自交和Cast自交的组织。用相同的单倍型分配步骤,85.7%的read可以正确分配到相关的基因型,而只有1.5%是错配的。
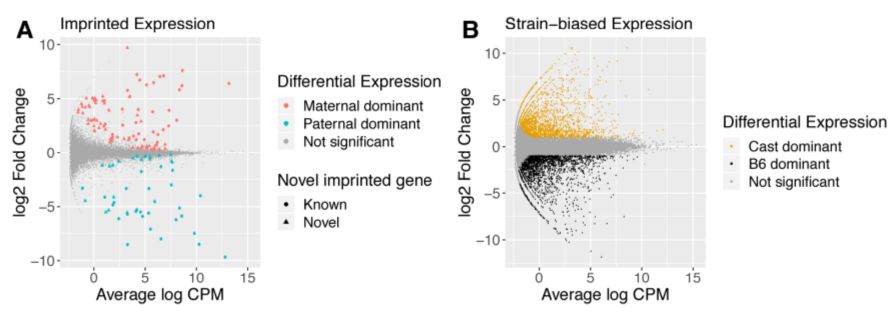
- 亲本特异和品系特异的基因表达
对B6和Cast正反交和自交的F1的胎盘组织分别进行RNA-seq测序。结果发现135个基因具有亲本特异表达,其中88个是母本等位高表达47个是父本等位高表达。而这135个基因中,有53个是已知的印记基因,这其中又有17个(包含Fkbp6, Smoc1/2, Gzmc/d/e/f/g, Zdhhc14 and Arid1b)在近期的研究中被认为是印记的。剩下的65个基因则组成了小鼠胎盘中候选的亲本偏好表达基因。
- Nanopore测序观测到的已知的印记控制区
作者结合nanopore read的单倍型和甲基化数据来比较亲本等位间的甲基化。发现Nanopore测序的数据非常容易观测出已知的印记控制区的DMR,且与RNA-seq和RRBS的等位特异情况一致。作者发现Nanopore的数据涵盖了大多数已知的印记控制区的差异甲基化,而这种甲基化差异往往会延伸到印记控制区外。
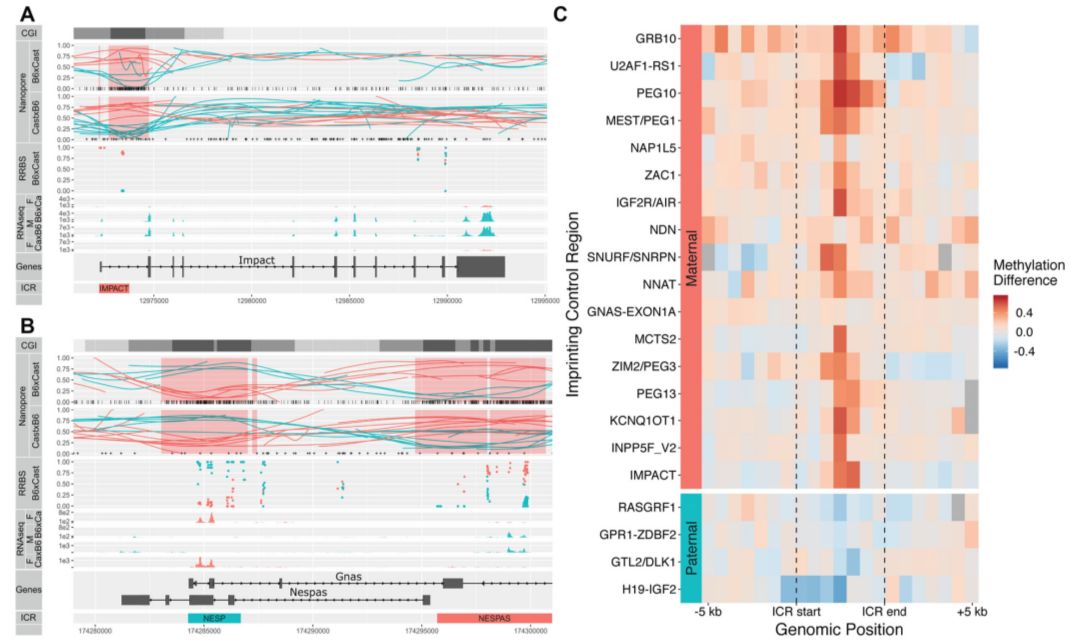
作者接下来尝试去发现亲本等位间的差异甲基化区域(DMR)以及品系特异的DMR。作者共得到933个DMR,其中309个可以被亲本的差异所解释,其余则是源自品系的差异。结合RRBS单倍型和RNA-seq的数据进一步证明了差异甲基化和差异表达,进而找到印记基因上感兴趣的DMRs。排名靠前的前20个DMRs中有15个对应于已知的印记控制区。虽然很多排名靠后的DMR可能是假阳性的,但是这些DMR依然包含了已知区域的印记表达。
- 差异甲基化分析上长read的优势
最后,作者通过几个例子阐述了相比于传统的重亚硫酸盐测序,基于nanopore的方法在识别亲本等位DMR上的诸多优势。这里,小编就不赘述啦。
参考文献
Gigante S, Gouil Q, Lucattini A, et al.Using long-read sequencing to detect imprinted DNA methylation. Nucleic Acids Research. 2019.02
如果您的项目有任何问题,欢迎点击下方按钮联系我们。








 京公网安备 11011302003368号
京公网安备 11011302003368号