之前跟大家分享了基因定位解决方案之联合分析,今天就跟精细定位的另一种方法,也就是我们精细定位常用的方法。
传统QTL精细定位的方法是利用高代回交群体(通常为NIL群体),也就是除了某几个片段来源于供体亲本,基因组其他区域来源于受体亲本,这将数量性状质量化,屏蔽遗传背景的干扰,如果研究微效QTL,这种方法也可排除主效QTL的干扰,但其构建时间长和构建过程复杂等特点使得科研工作者耗费过多精力在精细定位这个过程。
随着NGS测序成本的降低,使得基因组信息不断的公布,标记数目和种类不断的增加,简单易构建的初级分离群体也不再只限于初定位,其可结合定位区间内KASP标记进行高通量基因分型,进而加速精细定位进程。
接下来就回顾一下我们之前推送过的黄瓜耐涝SLAF-BSA文章(详细见《Plant Journal_SLAF-BSA助力黄瓜耐水淹机制解析》的大致内容,其中就利用KASP技术对F2大群体进行精细定位~~~
题目:The major-effect QTL CsARN6.1 encodes an AAA-ATPase domain-containing protein that is associated with waterlogging stress tolerance through promoting adventitious root formation
期刊:The Plant Journal
合作单位:扬州大学等
本研究利用SLAF-BSA技术,将黄瓜耐涝基因定位在301kb区间内(初定位群体为949株,选择50+50的混池),针对定位区间的SLAF标签的SNP信息设计KASP标记和dCAPS标记,并对6691个F2子代和6个F2:3家系进行分型,结合重组个体基因分型和表型数据,将ARN6.1定位在36.1kb的范围内,后期结合转录组,找出候选基因CsARN6.1。

图1 ARN6.1精细定位结果
除了传统的F2、RIL和NIL群体,剩余杂合系(RHL)群体也是一种适合于QTL精细定位的群体,其是利用分子标记可以直接从重组自交系(RIL)或回交自交系(BIL)群体中筛选出在目标区间内杂合而背景纯合的个体,通过自交构建成RHL群体。
同样地,我们简单回顾一下之前推送过水稻SLAF遗传图谱文章(详细见《继12连发,SLAF-遗传图谱再创佳绩》)的大致内容,其中就用到了RHL群体进行定位~~~
题目:TGW3, a Major QTL that Negatively Modulates Grain Length and Weight in Rice
期刊:Molecular Plant
合作单位:中国科学院植物研究所等
本研究利用SLAF-遗传图谱和粒形差异RIL群体表型结合定位到一个水稻粒形相关的noval 主效QTL——qTGW3。针对这个QTL构建了RHL群体,定位区间内开发标记对RHL群体检测,找到54个重组单株,结合表型将定位区间缩小到18.7Kb区域,其中含有3个候选基因,通过基因注释发现LOC_Os03g62500编码GSK3/SHAGGY-Like家族的蛋白激酶,已有报道此家族有成员与粒形相关,所以被锁定为候选基因。
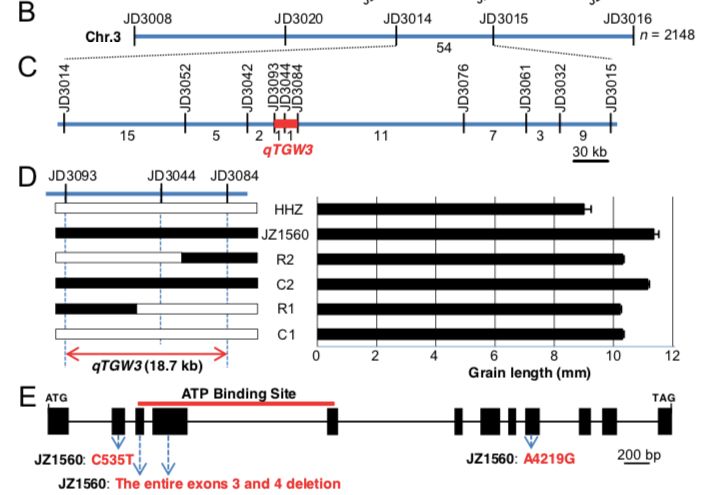
图2 qTGW3精细定位
结合上次呢,总共跟大家分享了常用的2类精细定位的方法,如果各位有补充的话,下面留言区等着你呦。
今天篇幅较短,主要总结一下常见精细定位的思路。主要强调了一下KASP技术和RHL群体在精细定位中的应用(虽然很常见,但还是忍不住磨叽一下),百迈客同样有KASP业务,如果您有需求的话,点击下方按钮联系我们。
参考文献:
Xu X, Ji J, Xu Q, et al. The major-effect QTL CsARN6.1 encodes an AAA-ATPase domain-containing protein that is associated with waterlogging stress tolerance through promoting adventitious root formation[J]. Plant Journal, 2018.
JieZheng Y, Ming M, Chen B,et al. TGW3, a Major QTL that Negatively Modulates Grain Length and Weight in Rice [J]. Molecular Plant,2018








 京公网安备 11011302003368号
京公网安备 11011302003368号