特异的SV是与疾病的易感性相关的,SV的区域通常包含疾病相关重要基因。许多癌症基因组有着显著的遗传变异,并且特异的SV被认为能够通过破坏基因结构,调节基因表达,创造融合事件或者产生基因拷贝数来促进肿瘤发展。不知道SV是什么的请到最下面看科普。
据统计,基因组结构变异可能导致的遗传性疾病已经超过1,000种,对于每个人来讲其基因组都有至少20,000个的结构变异,这些变异带来的影响或许比SNV或InDel还要大。然而,尽管SV的普遍存在且与癌症特殊关联,但是许多SV分类的分子组织及产生机制还不明确。这在很大程度上是由于当前技术(就是二代测序)无法发现具有高特异性的全谱SV。
据报道,短read方法缺乏敏感性,只有10%-70%的SV可以被检出,却有高达89%的错误发现率,且不能鉴定复杂嵌套SV带来的影响。
三代测序因其读长长,能够大幅提升SV的可靠性和分辨率。根据文章的结果和百迈客的实测数据总结起来,用ONT测SV至少要15X。
具体原因是什么呢?且听小编细细道来~~
Pacbio和ONT测序的长read能够大幅提升SV检测的可靠性和分辨率。平均10kb或者更长的read可以更准确的比对到重复序列上,这些可能介导SV的形成。长read更可能跨过SV断点。当然除了优势,长read也有新的挑战,Pacbio测序有10-15%的错误率,Oxford Nanopore 测序有5-20%错误率。因此急需一种新的SV检测方法,Sedlazeck F J 等人开发了Sniffles软件。
根据两个人类数据集的错误情况和read长度,作者对两条人的染色体模拟了50X Pacbio 和ONT read 。纯粹的统计分析发现,近10X覆盖度的数据(平均长度10kb)就足够去推断所有SV断点(一瞬间觉得自己可以省好多钱有木有),然而对于100bp的短read双端测序至少要25X覆盖度。当前这个统计只是一个理想情况,比如缺乏了重复和覆盖度的偏移,因此是低估了所需的覆盖度的。
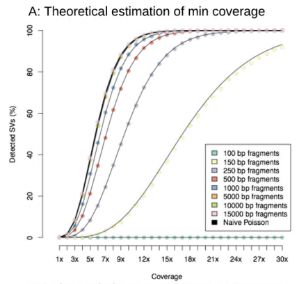
理想很丰满、现实很骨感!理想情况下用10x覆盖度测三代read就能检测出来所有的结构变异,但是现实肯定不够啦~ 作者对真实的Pacbio 55X数据,和Nanopore 28X数据所检测到的SV和低深度下所检测的SV进行比较。对于Pacbio数据,15X的时候对于NA12878和SKBR3样品的SV能识别到69.64%和67.24%,如果提升到30X时,可分别识别到80.05%和76.63%。SKBR3的识别率相对较低主要因为它是癌症样品,有些极端的拷贝扩增。所以癌症样品要想识别到更多更准的SV,需要适当提升测序深度。
对于Nanopore的数据,在20X的覆盖度时就能达到82.24%的准确率和84.23%的识别率。不过这可能是因为ONT数据只测了28X。
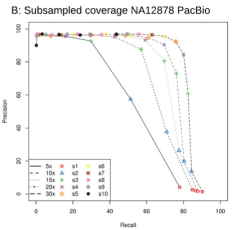
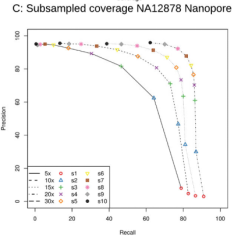
尽信书不如无书,小编本着对科研(领导)的认真态度(“逼迫”),对公司的一正常人的血液进行Nanopore DNA测序(测序深度为40X)识别SV,随机抽取不同深度下的数据量5X,10X,15X,20X,30X使用相同的参数进行SV识别,合并所有样品的SV,对每个样品进行强制重新识别SV。以40X数据在支持read数大于10下所检测出的SV为金标准,判断低深度下所能检测出的SV情况,如下表:
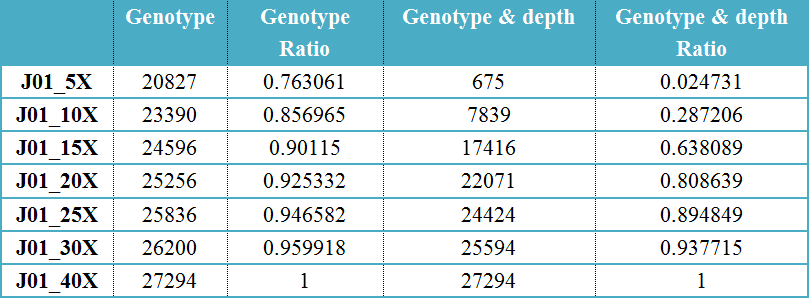
注:Genotype列代表不同深度下识别出的和40X SV基因型相同的SV个数,Genotype ratio为SV占40X SV的比例。 Genotype & depth 为与40X SV基因型相同且read支持数大于10的SV个数,Genotype & depth Ratio为基因型相同且read支持数大于10的SV比例。
其实从结果上可以发现即使只用5X测序深度的数据也能够识别出很高比例的SV,但是如果考虑到支持的read数,所能识别出的SV比例就瞬间少了很多。其实也能理解啦,毕竟深度在那里呢~
所以,依小编愚见,15X数据的结果相对还是可以的,不过该测试数据是妥妥的正常人呦,如果癌症样品还是建议再多测一些呢~
参考文献
Sedlazeck F J , Rescheneder P , Smolka M , et al. Accurate detection of complex structural variations using single-molecule sequencing[J]. Nature Methods, 2018.








 京公网安备 11011302003368号
京公网安备 11011302003368号