2018年9月18日,国际知名期刊BMC Biology在线发表了沈阳农业大学水稻研究所徐铨老师关于水稻产量和质量遗传位点研究的文章,该文章不仅解析超级稻品种“沈农265”的基因组,同时也分析了在不同生态环境和不同遗传背景下功能基因的功能变化,同时鉴定了多个候选基因,并使用CRISPR/Cas9基因编辑技术验证了基因的功能,该结果将会促进不同稻区籼粳稻杂交聚合亚种的遗传研究。百迈客有幸参与完成了高密度重测序遗传图谱的构建和QTL定位等工作,为后续基因挖掘奠定了坚实的基础。
英文题目Genome sequencing of rice subspecies and genetic analysis ofrecombinant lines reveals regional yield- and quality-associated loci
出版期刊: BMCBiology
五年影响因子:7.55
发表年份: 2018年
合作单位:沈阳农业大学水稻研究所
实验材料
试验设计:3个种植环境(沈阳、江苏、深圳),2年(2015-2016)
研究方法
测序:二代HiSeq2500,三代SMRT
分析:重测序遗传图谱,转录组测序,基因注释
实验验证:CRISPR/Cas9
研究背景
籼稻和粳稻为亚洲栽培水稻的两个亚种,它们的地理分布以及形态学性状差异明显,揭示其农艺性状背后的遗传基础对于水稻生产非常重要,然而,其遗传背景、生态条件以及农艺性状之间的关系尚不清楚。沈农265(SN265)是我国第一个商用的超级稻,它作为中国北方骨干亲本引领着育种方向。本研究利用二代和三代测序对北方粳型超级稻SN265和R99进行测序,并获取SN265高质量基因组,又构建了RIL群体重测序遗传连锁图谱,并对多个重要农艺性状进行了QTL分析。此外,还发现若干基因功能随生态环境的变化而改变,并且许多等位基因对不同的遗传背景呈现差异性的响应。
主要结果
对RIL群体及其亲本进行全基因组重测序,每个RIL系测序深度为6.25×,亲本R99与SN265测序深度分别为30×与32×。通过SOAP进行比对和SNPcalling,共得到1,708,775个SNP,过滤低质量后得到1,456,445个高质量的SNP;利用划bin策略进行图谱构建,得到3569个bins,平均长度为58.17 kb(图1);使用百迈客作图软件HighMap进行图谱构建,总图距为1965.33 cM,平均图距仅为0.55cM,与参考基因组共线性分析分析显示,最小相关系数为0.9725
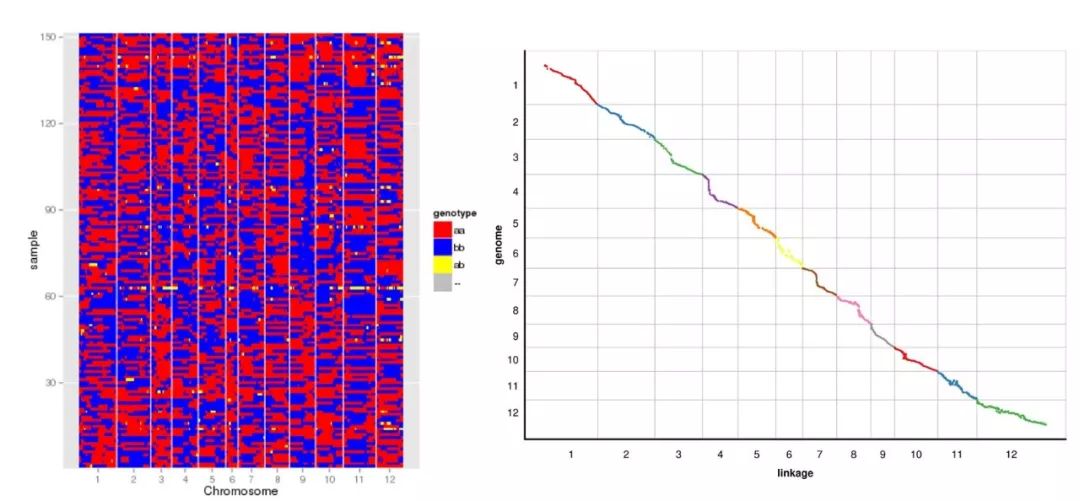
图1 利用重测序SNP构建的bin图和遗传图谱与参考基因组共线性分析
为了填补北方超级稻高质量基因组序列的gaps,作者对SN265基因组进行了de novo测序和组装。利用P5-C3 SMRT测序,获得24.94 Gb的原始数据,平均读长为9.6 kb,二代测序共获得22.05 Gb的原始数据,组装得到SN265的基因组,大小为364.45 Mb,contig N50 达到6.96 Mb,通过从头预测、同源预测以及RNA-seq分析后共获得37,609个基因(图2)。对R99进行低深度(30×)三代重测序和基因组组装,获得了R99389.6 Mb的基因组,contig N50也达到3.05 Mb(图2)。
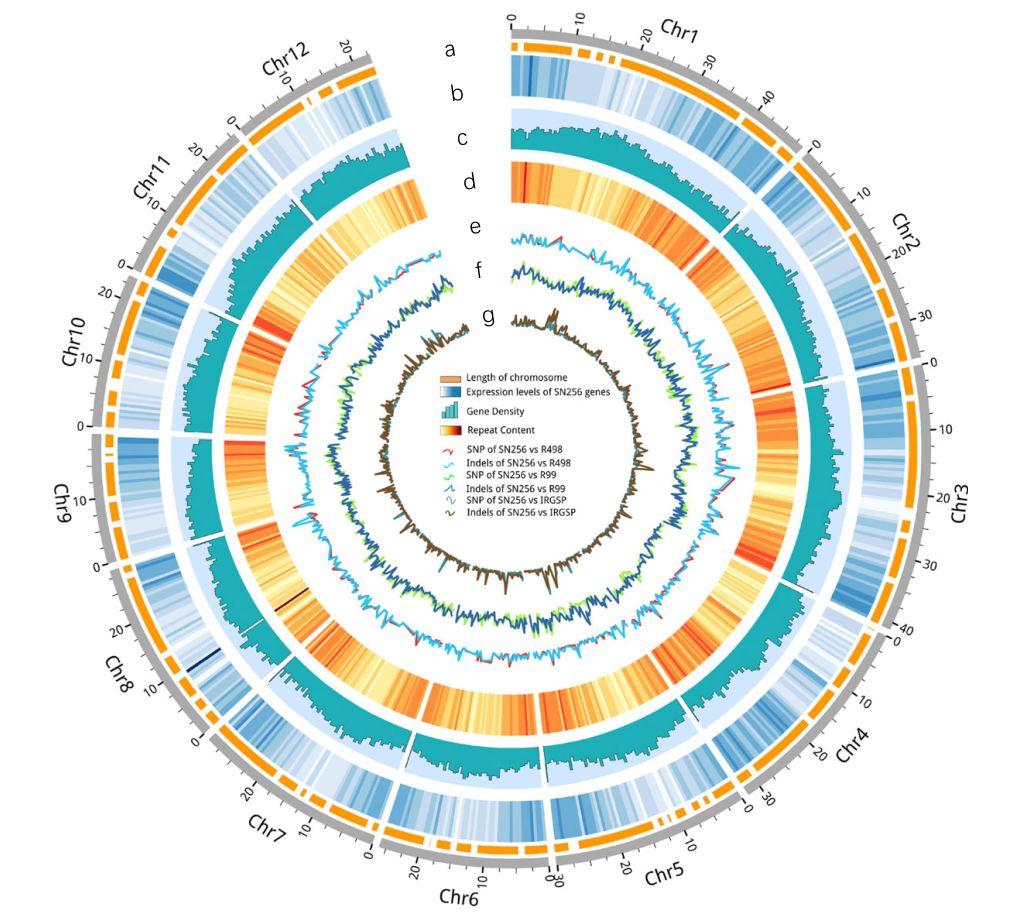
图2 亲本基因组组装结果展示
为了阐释遗传背景与农艺性状的关系,作者以粳稻和籼稻亚种间特异SNP计算得到每个RIL系的籼型血缘百分比。结果显示,籼型血缘百分比主要影响穗长与粒形(图3)。
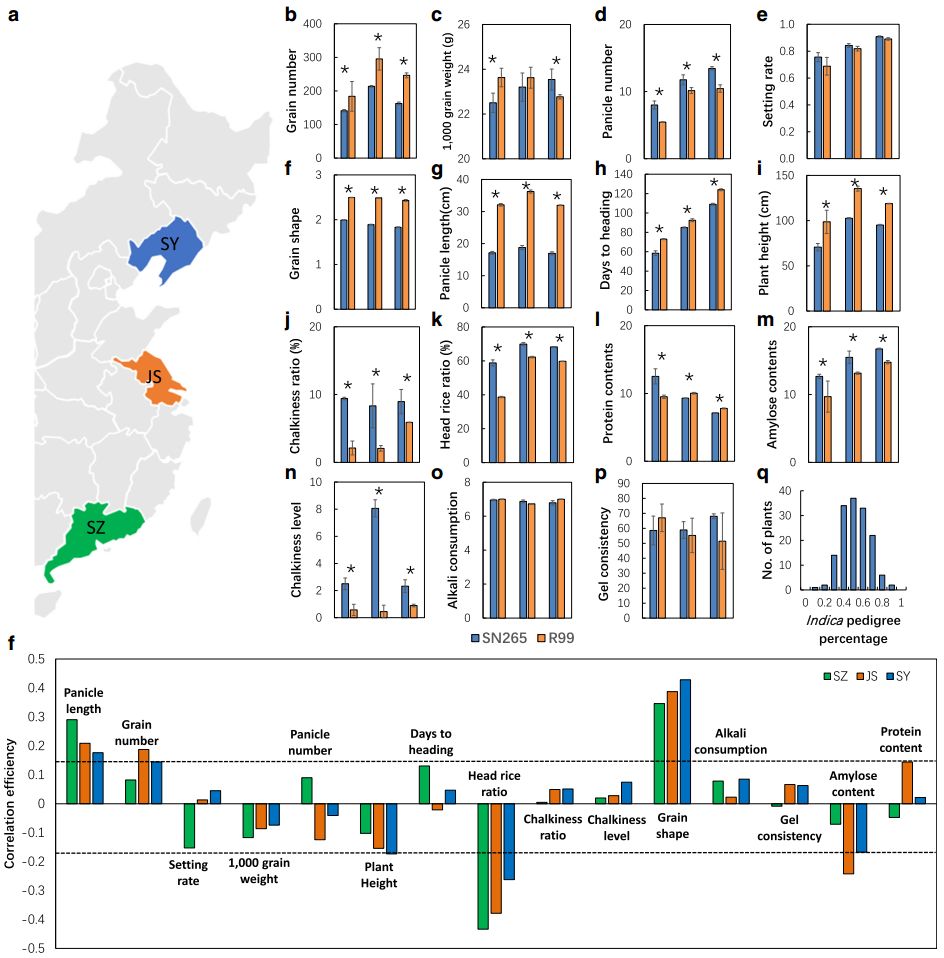
图3 不同籼稻型血缘比例的RIL子代在不同环境下农艺性状表现
通过收集三个地点15个产量与质量相关的表型数据,进行QTL的挖掘,结果共检测到79个QTL,许多QTL呈现成簇分布的特点,暗示多种性状可能由一个基因或多个紧密连锁的基因控制。有些QTL在3个环境下均被检测到,另有一些位点仅在1个环境下被检测到。
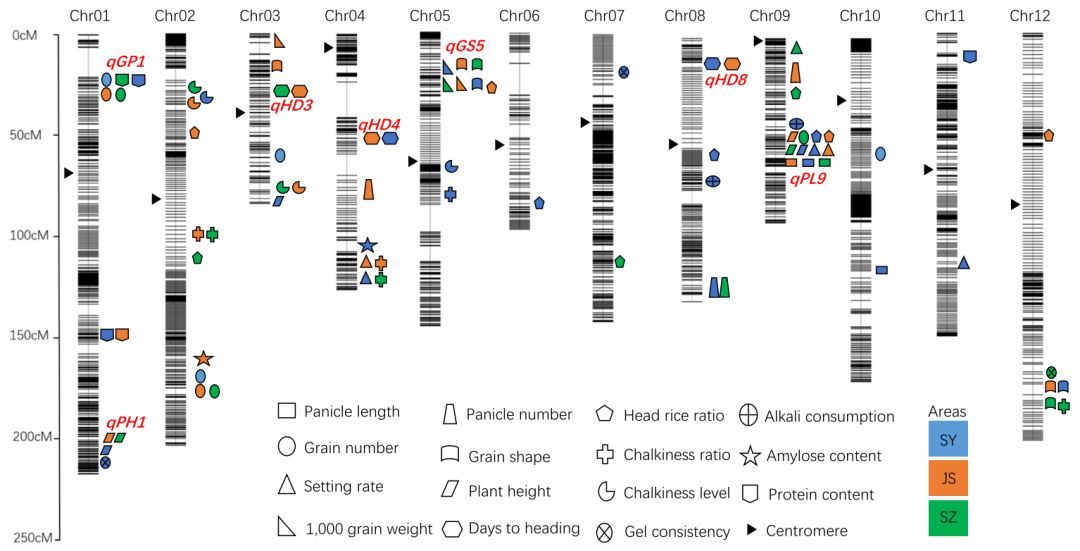
为了确定QTL簇的候选基因,进一步做了基因的精细定位。首先,关注的是chr9上的 qPL9簇,候选基因被定位在43kb的区间内,其中包含7个注释基因,通过序列比对分析发现一个候选基因DEP1上的exon5中存在碱基序列的替换,即R99中637bp的片断在SN265中被替换成12bp的序列;接着共分离分析发现SN265型的dep1具有明显短于R99的穗长(图5)。为了证明DEP1是qPL9的候选基因,利用 CRISPR/Cas9技术对Sasanishiki品种中的DEP1基因诱变,获得五个突变体株系,其中四个突变体发生移码突变,呈现短穗长,另外一个突变体产生两个氨基酸的替换,其穗长与Sasanishiki无显著性差异(图5),暗示DEP1是qPL9簇中控制穗长的基因。
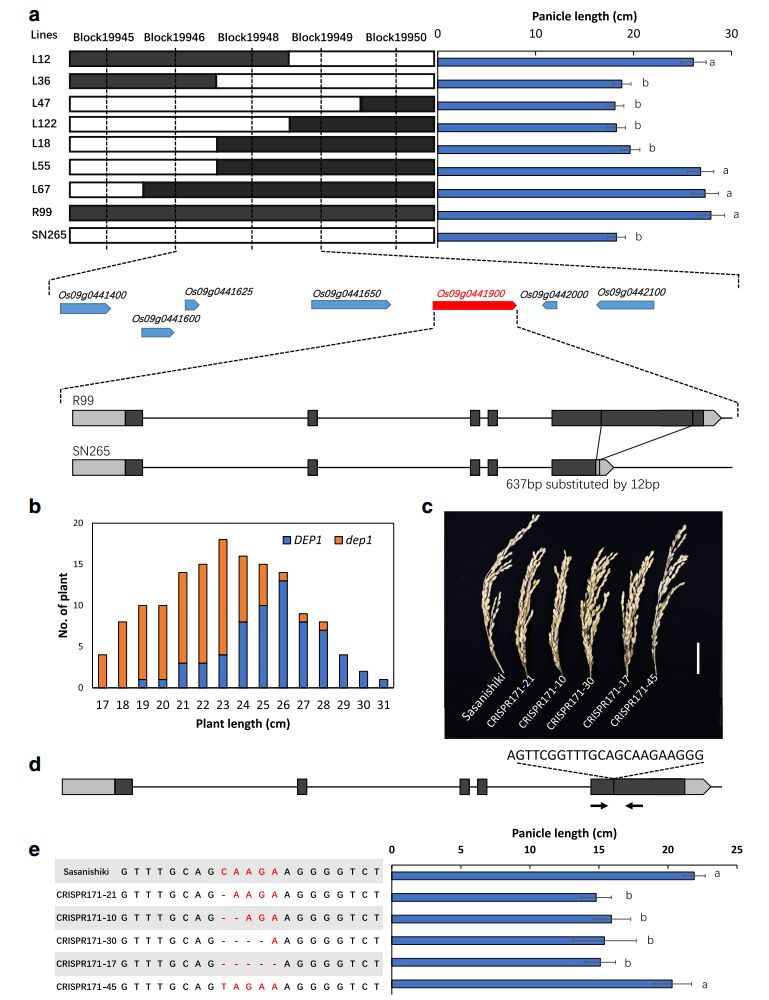
图5 DEP1精细定位及CRISPR/Cas9突变结果
接着在Chr1短臂上的qGP1簇上定位到候选基因Gn1a,在长臂定位到SD1为控制株高的候选基因;在Chr5上鉴定到的GW5上游存在1212bp序列的缺失;在qGS12簇中的Os12g0610600存在一个SNP,负调控水稻耐旱反应。而且,基因GW5与qGS12的不同组合形式会显著影响水稻种子的粒型。此外, DTH8、SDG708与phytochrome B (PHYB)分别位于Chr8、4、3上,是控制抽穗期的候选基因。
为了揭示生态环境与遗传背景对基因功能的影响,首先,作者发现Gn1a对水稻单穗籽粒数目的贡献主要集中在江苏与沈阳两个实验点,而DEP1仅仅在深圳被检测到是控制单穗籽粒数目的QTL(图6)。对以上三个种植区,又进一步比较携带有 DEP1/dep1与Gn1a/gn1a的不同水稻材料,结果发现在高纬度生态区dep1对单穗籽粒数目的贡献减少,而gn1a对其贡献明显增强(图6)。类似地,DTH8、 SDG708与PHYB在江苏均被检测到,而PHYB在深圳也被检测到,同样, DTH8、 SDG708在沈阳也被检测到;结果显示DTH8、SDG708几乎不会影响在深圳的抽穗期,PHYB在沈阳有较弱的作用(图6)。概括起来,Gn1a、 SDG708、SD1与GW5的功能在高纬度地区被增强,另一方面,PHYB与DEP1的功能随着纬度的北移被破坏,有趣的是,DTH8在中纬度地区具有较强的功能,纬度的南移或北移都会削弱其功能。
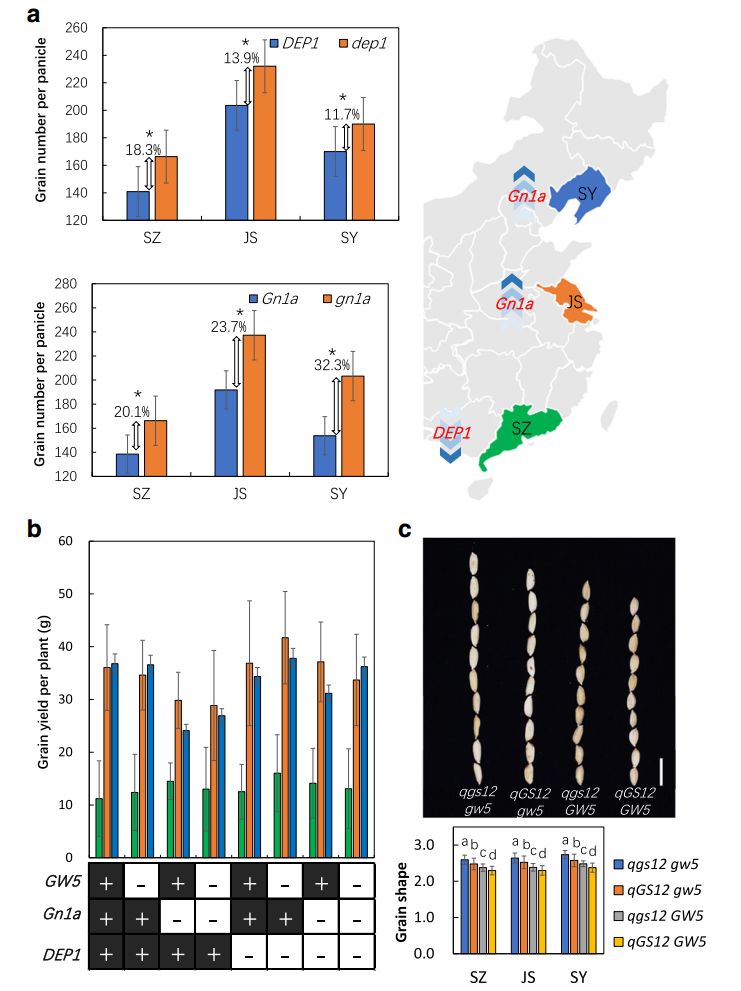
图6 不同生态环境与不同遗传背景下基因表现的差异
为了揭示遗传背景对基因功能的影响,根据籼型血缘百分比将RIL群体分为三组:粳型、中间型与籼型。经分析发现,不同遗传背景的基因功能也不同,例如,粳型SD1随着籼型血缘百分比的增加,株高降低,而籼型sd1随着百分比的增加,株高增加。又比如,在深圳,粳型PHYB随着籼型血缘百分比增加延迟抽穗期,而在江苏与沈阳加快抽穗期。此外,在三组群体中,中间型的农艺性状表现最差,例如,在DEP1与Gn1a遗传背景下,中间型拥有最低的单穗籽粒数目。
结论
本研究为水稻育种功能基因的利用提供信息,即合适的生长环境与遗传背景对基因功能的影响;北方粳稻品种沈农265基因组参考序列的发布为植物科学家与作物遗传改良提供宝贵资源。
1、重测序遗传图谱为研究打下坚实基础。
2、不同稻区多年的表型数据,定位到了多个QTL簇,从而将研究重点放在这些QTL簇上。
3、结合3代和2测序获得了沈农265高质量的基因组序列。
4、独辟蹊径地阐述了基因在不同环境和不同遗传背景下功能的转变,这也是本文最大的亮点。
欢迎与我们的生信工程师沟通,我们将免费为您设计文章思路方案








 京公网安备 11011302003368号
京公网安备 11011302003368号