花生作为我国重要的经济作物,广泛种植于热带和亚热带地区,是提供重要的蛋白和油料的基础。作为豆科的重要分支之一,花生属一共包括30个二倍体品种,1个异源四倍体野生花生(A.monticola)和1个异源四倍体栽培花生(A.hypogaea)(2n = 4x = 40)。作为栽培花生农艺性状改良的重要野生资源供体,野生四倍体花生的基因组也一直是国内外学者的研究热点。成功破译四倍体野生花生的基因组有助于科学家和育种专家对A.hypogaea起源及驯化过程的理解。河南农业大学殷冬梅教授团队与北京百迈客生物科技有限公司、中国科学院等多家单位联合攻关,成功破译复杂的异源四倍体野生花生基因组密码,相关成果于2018年6月19日发表在GigaScience上:“Genome of an allotetraploid wild peanut Arachis monticola: a de novo assemble”。
该团队充分考虑野生花生中高度同源的异源四倍体基因组的复杂性,充分利用SMRT + Hi-C + IRYS + Illumina等测序平台技术优势,采用基因组组装技术成功破译了四倍体野生花生基因组,最终得到染色体水平的高质量野生花生参考基因组。组装基因组大小为2.62Gb,为预计基因组大小的97%,contigs N50和scaffolds N50分别为106.66Kb,124.92Mb,其中96.07%的序列挂载到20条染色体上,91.83%的序列可以确定顺序和方向。野生花生基因组的发布对于理解花生属和豆科作物进化具有重要的科学价值,促进花生以及其他油料作物的功能基因组学发展和分子育种。

野生异源四倍体花生基因组的难点
通过K-mer分析预测该基因组的大小为2.42Gb(实际大小约为2.7Gb),重复序列为75%,基因组杂合度为0.1%。通过2015年发表在Nature Genetics上的2个祖先种序列可知A和B两套亚基因组的同源性高达93%,属于高度同源的异源四倍体基因组,甚至同源性超越了某些同源多倍体的基因组。
该基因组的难点不是仅仅获得一个高的Contig N50指标,而是能够将四倍体的序列信息组装完整,并进行区分。因为亚基因组的高度同源,又具有75%的高重复序列这给组装的完整性带来极大的困难。该基因组的杂合度仅有0.1%,低杂合对于二倍体物种来说是有利于基因组组装的,但其对于多倍体物种,非常不利于亚基因组的区分(因为两套基因组序列太类似,甚至连SNP都很少)。
百迈客对野生异源四倍体花生的组装策略
在这项研究中,研究人员以野生四倍体花生A.monticola为研究材料,进行测序得到36X SMRT subreads + 76X HiC data + 210X Bionano Irys data + 50X Illumina reads的测序数据,整合多种组装工具的优势,最终获得了参考基因组水平的高质量组装结果,如下所示:
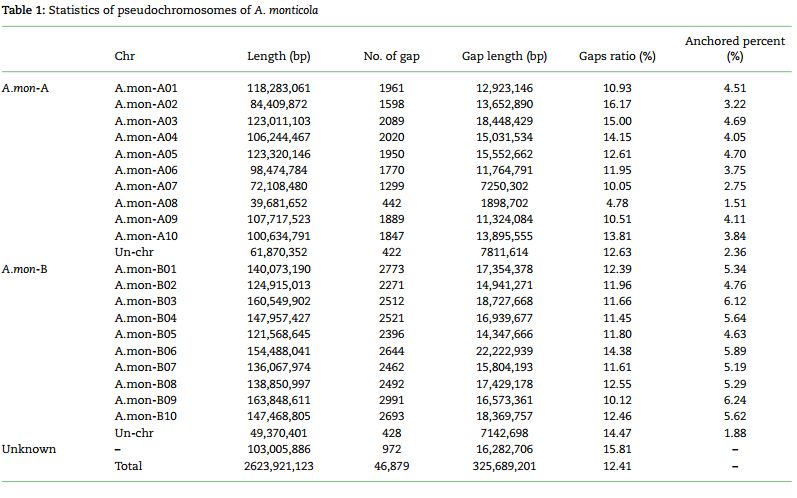
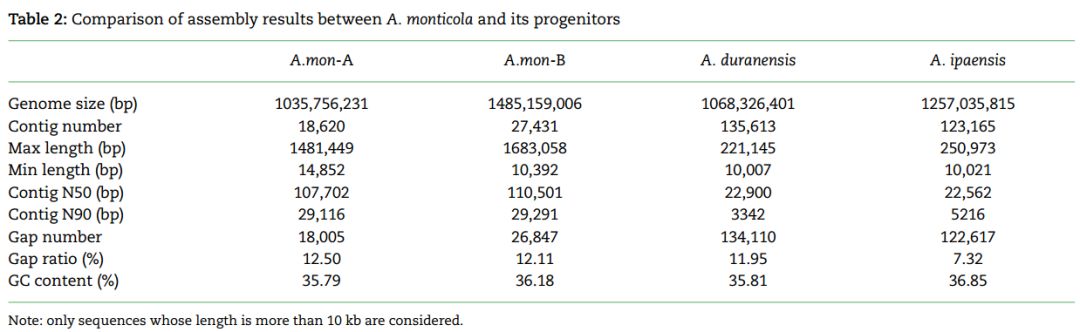
最终组装出2.62Gb的基因组序列,保证组装的完整性,又利用BioNano和Hi-C等方法对基因组进行区分最终A.monticola得到的subgenome与祖先A基因组A.duranensis、祖先B基因组A.ipaensis之间的比较如表2所示,野生花生A and B subgenomes与对应的基因组大小相近,基因组的完整性和连续性都有了显著提升,为后续的功能基因组研究打下了坚实的基础。
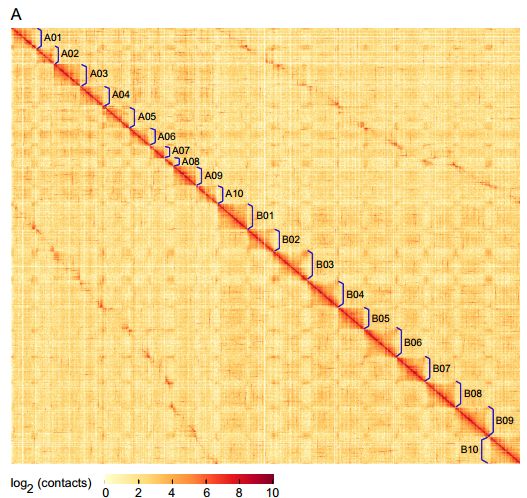
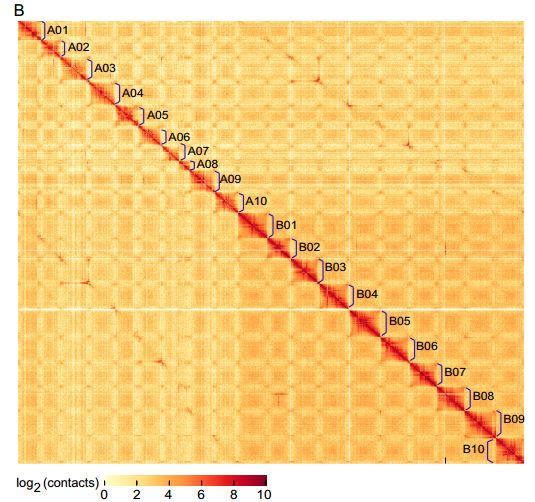
百迈客对野生异源四倍体花生组装的Hi-C热图评估
除了常规基因组的评估手段之外,我们还利用更直观的Hi-C对该基因组序列进行准确性评估,并与其祖先种A和祖先种B进行了比较。比较栽培种A.monticola得到的subgenome与祖先A基因组A.duranensis, 祖先B基因组A.ipaensis的Hi-C热图,整体热图一致性非常高。(A图是A.monticola的HiC热图,B图祖先A基因组A.duranensis, 祖先B基因组A.ipaensis的Hi-C热图)
该研究整合使用SMRT subreads + HiC + Optical data等多种测序手段并开发了一套全新的denovo组装策略,最终获得染色体级别的高质量的异源四倍体基因组,相比较于之前发表的基因组,contigs N50有了将近5倍的提升,并且包含了97%的野生花生的基因组序列。此项研究表明这种全新的组装策略对于异源四倍体基因组的组装是可行的,并且这也是野生花生基因组公布,对于研究祖先二倍体花生与栽培四倍体花生的进化,起源等研究起到了”桥梁”的作用。该项目得到国家基金委、河南省产业技术体系、河南省科技厅等诸多项目的资助。
针对这篇文献,您有哪些想法与我们交流?我们将免费为您进行文章思路设计。








 京公网安备 11011302003368号
京公网安备 11011302003368号