
目前,对基因表达调控的研究主要是以基因及其调控元件的线性关系为基础,然而,基因不仅仅以简单的线性形式存在,越来越多的证据表明染色质之间的相互作用在基因表达调节方面也起重要作用,即基因的表达调控存在三维空间网络,基因表达可被远程调控元件所调控。
基于3C的技术方法产生了大量的全基因组相互作用数据。本文(发表于2013年Nat Rev Genet)简述了主要的实验方法,更多篇幅用于描述最近开发的染色质互作数据集的分析、计算和建模方法。在此讨论3种方法:第1种方法,其目的仅仅在于识别比预期更频繁互作的一对或一组基因座,比如染色质环loop或特定的共定位事件,分析基因座间互作频率以鉴定更高阶的染色体结构域;另外2种方法分别为距离约束建模(restraint-based modeling)和聚合体(polymer)建模,使用包括基线互作和非特异性互作在内的所有互作数据来构建染色体空间模型的集合。之后,3D模型可以用来鉴定染色体空间组织的更高阶结构特征和DNA元件,以估计折叠过程中细胞内的染色质动力学以及细胞间的可变性。在此,作者讨论了这些方法的应用,包括如何确定染色体空间组织的原理,揭示新的染色质结构,并将这些结构与基因表达和调控联系起来。
一、实验技术——染色质构象捕获及其衍生技术
染色质构象捕获(chromosome conformation capture, 3C)技术原理是:(1)利用甲醛固定细胞核内的相互作用的染色质位点;(2)利用限制性内切酶将DNA切成片段状;(3)再用DNA连接酶对片段末端进行连接,从而捕获含有相互接触DNA片段;(4)利用PCR 或者测序的方法检测DNA片段的连接位点,获得染色质不同位点相互接触的频率;(5)数据分析,推断出染色质的空间位置信息,从而得到染色质相互作用位点的图谱。
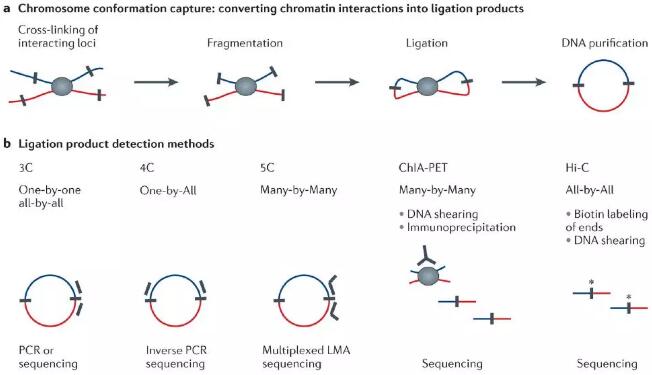
不同的3C衍生技术的区别在于捕获的连接片段检测和定量方式:
3C:经典的3C实验中,通过基因座特异性引物PCR检测单个连接产物,大多数3C通常仅能分析几十到几百Kb染色质之间的相互作用,通量低,费时费力——one vs one;
4C(“circular 3C”或“3C-on-Chip”):使用反向PCR产生单基因座的全基因组相互作用图,研究已知DNA片段(bait)与全基因组未知DNA片段之间的互作——one vs all;
5C(Chromosome conformation capture carbon copy):基于3C的基本原理,结合连接介导的扩增 (ligation-mediated amplification,LMA)来增加3C检测的通量,识别两组大量位点之间并行的数百万个相互作用,例如一组启动子和一组远端调控元件之间的互作——many vs many。
Hi-C(High-throughput chromosome conformation capture):用于对整个基因组所有位点间进行无偏差的作用分析的3C衍生技术,该技术有一个独特的步骤,即限制性酶切消化后用生物素标记的核苷酸补平缺口,有助于选择性纯化用于测序的连接产物。Hi-C提供了一个真正全基因组范围的相互作用图谱(该图谱的分辨率取决于测序的深度,常规测序数据量,即几亿reads时,小鼠或人类基因组中的染色质互作的检测分辨率为100Kb)——all vs all。
此外,还有将3C与染色质免疫沉淀结合,以研究与特定蛋白结合的基因座之间互作的技术:
ChIP-loop(chromatin immunoprecipitation-loop assay):常见的是ChIP-3C,以过量的限制性内切酶将染色质-蛋白质交连物酶切消化后,用所研究蛋白质的特异抗体进行免疫沉淀,然后再连接酶切产物,后续步骤和3C相同——one vs one;
ChIA-PET(chromatin interaction analysis by paired-end tag sequencing):对感兴趣的蛋白质结合位点之间的远程互作进行全基因组分析——all vs all。
二、研究染色质的空间组织形式
源于成像技术的见解
利用各种改进的成像技术进行的详细研究揭示了染色体在整个细胞核内的几个组织原则:
1)在许多生物体的间期细胞中,染色体不易混合,而是占据它们自己的独立区域;
2)染色体区域接触的地方,可以形成交织的区域,为位于不同染色体基因座之间的潜在功能性互作提供机会;
3)转录事件在整个细胞核中并非广泛地发生,而是发生在富含RNA聚合酶Ⅱ和转录以及RNA加工的其它组分的区域。这意味着积极转录的基因倾向于共同定位;
4)基因组的转录失活片段也倾向于彼此相关联,并且常常位于核周边、核仁周围或果蝇中的亚核结构上,如多梳体。
这些发现表明细胞核在空间和功能上可以划分不同区域,基因座的亚核定位与基因表达相关。
染色质构象捕获及其衍生技术(3C-based technologies)
成像技术的缺点:不易全面分析完整基因组的三维折叠,且分辨率达不到Kb水平。
基于3C及衍生技术,克服了成像技术的缺点,能够以足够的分辨率在全基因组范围内研究染色体折叠,以及涉及的基因和调控元件。详细介绍见上述实验技术——染色质构象捕获及其衍生技术。
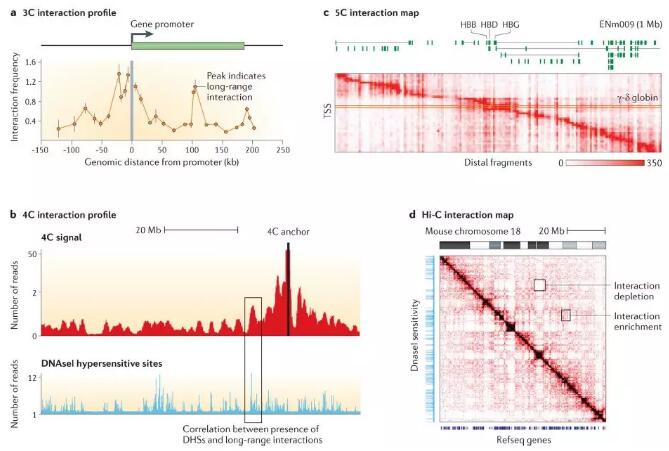
3C、4C、5C和Hi-C数据集互作图示例:
三、解析染色质互作数据
这些3C及其衍生技术报告了细胞群体中两个基因座空间上紧密接近的频率,但未区分功能性与非功能性的位点间关联,也未揭示导致其共定位的机制。空间上紧密接近包括以下几种情况:1)Direct interaction:两个基因座之间直接、特异性接触的结果(由结合它们的蛋白质复合物介导);2)Interaction with the same sub-nuclear structures:成对基因座与相同亚核结构间接共定位的结果(例如核纤层,核仁、或转录工厂等)。3)Bystander interaction:在某些细胞中,由于邻近的某些远程互作或其他约束因素决定的染色质纤维的堆积和折叠的造成的非特异性接触,或者由于拥挤核中的随机(非特异性)碰撞导致的非特异性接触。4)Baseline(polymer) interaction:染色质纤维非常长,而且柔韧,染色体具有聚合体性质,因此,即使在没有任何特定高阶结构的情况下,这个特征在很大程度上也决定了基因座间相互作用的频率。
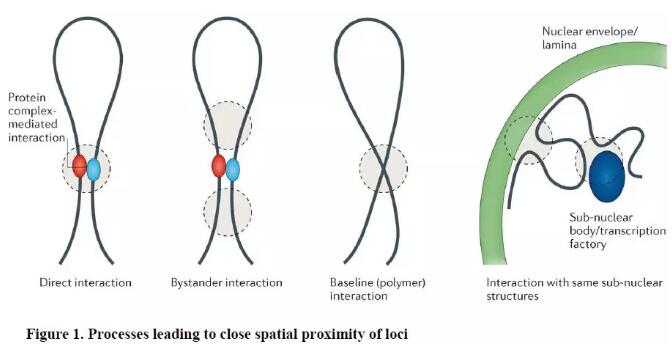
染色质纤维的√确三维结构在其它同类细胞之间甚至是高度可变的,并且在细胞内局部区域(大约Mb大小)是动态的。这解释了为什么全面的染色质互作数据集通常显示一个基因座几乎与基因组中的任何其它基因座具有互作的概率。检测到的每个染色质互作或连接产物实例,表示群体中单个细胞中的一对基因座的互作。因此,3C互作频率数据代表细胞固定时,存在空间上紧密接近的基因座所在的那部分细胞,并且只有在基因组折叠显示出巨大的细胞间异质性时才能解释该数据。这些突出了全面染色质互作数据集的复杂性质:互作数据表示大量细胞群体间相互作用的总和,并且在每个细胞中染色体构象由作用于染色质纤维的许多不同约束决定。
目前,分析染色体构象的挑战正在从开发用于生成日益全面和定量数据集的实验方法转变为构建分析工具以解释相互作用数据。 作者阐述的第一种方法是用来识别点对点的成环互作,例如, 启动子和基因调控元件之间的互作。
四、定位调控元件的靶基因
鉴定成环互作
后生动物基因组中,每个基因被大量元件包围。一个主要问题是:决定特定时间哪些元件调控特定基因的原理是什么。基于最近十年的单个基因的详细分析,以及最近更全面的全基因组范围的研究报道发现,调控元件与其靶基因进行通信的主要机制是通过染色质成环(chromatin looping),这使得线性距离很远的基因座可以在空间上密切接近。
单基因座研究中,经典的3C技术被用于检测感兴趣的元件之间的相互作用频率,例如,启动子和延伸至数百Kb的侧翼染色质间。分析这样的“锚定”(anchored)互作图,可以找到比预期更频繁地与锚定位点互作的远侧基因座,也就是成环互作(loop interaction)。通常,相互作用频率随着基因组距离的增加呈指数衰减。许多研究中,loop互作指在整体衰减基线之上观察到局部峰(peak)。3C分析本质上是定性,并且基于互作图的简单视觉检测来识别交互频率中的peak。比较不同细胞或不同条件下获得的互作特征,可以提供更多信息,包括统计定量以及当远程互作是条件性或细胞类型特异性时的loop互作信息。
特定基因组成环互作示例
经典示例之一:基因座位控制区(locus control region,LCR)和相距40-80Kb的一组远端β-珠蛋白基因之间的长程互作。小鼠和人类的3C研究检测到珠蛋白表达细胞中这些元件之间的显著互作,且这些互作在不表达这些基因的细胞中显著较不频繁(如大脑)。这些互作由特定的转录因子介导,包括结合LCR和基因启动子的EKLF1和GATA1。此外,成环互作通过促进RNA聚合酶Ⅱ的募集和磷酸化直接促进转录。染色质成环构成基因调控元件在基因组远距离调控基因的常见机制。
五、综合分析Loop
5C数据loop分析
5C技术通过并行绘制多个基因座之间的互作图谱,允许更全面地分析大量基因的染色质成环互作。例如,最近一项研究中,绘制了3种人类细胞系超过600个基因启动子的互作图谱,分辨率:单个限制性片段(〜4Kb)。假设大多数交互不是特异的loop交互,从整个数据集中估计互作频率的基线值,由此估计出各基因组距离的互作频率基线。然后通过检测显著高于该基线的信号,以选定的p值和错误发现率鉴定loop互作。与经典的3C单基因座对研究相比,这种方法统计学分析上更严格,可以鉴定该基线上的显著peak。示例见下图。
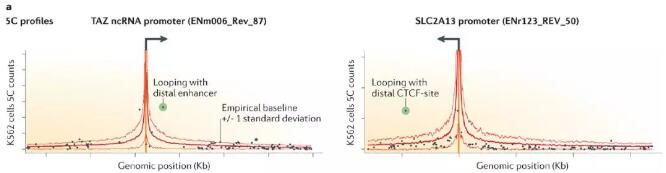
5C的缺陷:1)受限于用于定义预期互作频率的模型和假设;2)被检测的细胞群体中的实际互作频率(发生loop互作的细胞比例)仍然是未知的,并且可能非常低,这使得很难评估这些相互作用在任何给定细胞中的功能作用。
关于loop景观的见解
尽管5C技术存在上述缺陷,但仍旧揭示了染色质不同区域间远程互作参与基因表达调控的普遍规律。Sanyal等人发现了基因启动子和远端基因座之间有数千个重要的远距离loop互作,强调了许多基因启动子通过染色质环与远端元件互作的观点。普遍规律如下:1)许多染色质成环事件是活性基因启动子和类似于活性增强子的远端元件之间的细胞类型特异性互作,这与这些染色体结构在基因活化中的作用一致;2)其中一类丰富的远程互作即是启动子区与绝缘蛋白CTCF结合位点之间成环;3)通常认为调控元件可以调节*邻近的基因,但是成环互作经常跳过一个或多个基因,这表明基因和元件的线性排列是它们之间功能和结构互作的较差预测因子;4)基因和调控元件之间的关系并不唯一:一个基因可以与多个远端元件互作,同时一个元件也可以与多个基因互作。
此外,研究发现启动子周围的成环互作模式不对称:启动子可与位于转录起始位点上游或下游的远端元件互作,但成环互作常见于上游〜120Kb。不对称原因尚不清楚,但是可能暗示某种方向性。从这些研究可以看出,染色体是由远距离互作驱动的高度复杂的三维网络。同时提出了新的问题:介导它们的蛋白质以及这些成环互作如何促进基因调控。
六、拓扑相关结构域(topologically associating domains, TAD)
5C和Hi-C等技术,以不偏倚的方式对感兴趣的基因组区域或整个基因组中的所有相互作用进行分析,从而鉴定染色体的结构特征。后生动物基因组的一个突出特征是形成各种类型的染色体结构域。果蝇、小鼠和人类染色体的研究发现,染色体由离散的拓扑相关结构域(TAD)组成,其大小为数百Kb(见下图中每个绿色横线对应的三角形,TAD染色质局部相互作用较为强烈的一个作用单元)。
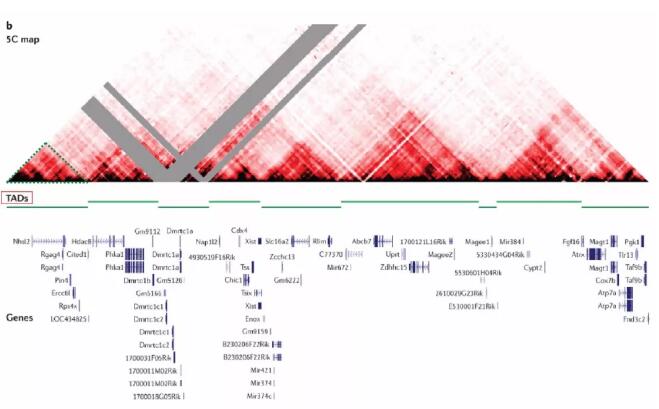
包含小鼠X染色体失活中心的4.5Mb区域的高分辨率5C互作图揭示了一系列大的结构域。位于这些TADs内的基因座往往频繁地相互作用,但它们与位于其领域之外的基因座的相互作用要少得多,即TAD内部的相互作用强,不同TAD间的相互作用弱。这种特征使研究人员能够通过分析分辨率较低的全基因组Hi-C互作图与隐马尔可夫模型方法结合,来识别整个人类和小鼠基因组中的TAD。TADs是染色体的通用结构模块,人类和小鼠的基因组都由2000多个TAD组成,覆盖了90%以上的基因组。
TAD是由遗传编码的边界元件定义的。删除X染色体失活中心中两个TAD之间的边界,导致两个侧翼TAD的部分融合(并非完全融合),这表明激活了一个新的边界。全基因组研究发现TAD边界富含CTCF结合位点,尽管CTCT也经常结合TAD内部基因座。TAD的边界除了富集 CTCF的结构域,还有大量的持家基因、tRNAs、SINE 反转录转座子等 DNA 元件。建立TAD边界的机制仍不明确。
(CTCF, CCCTC binding factor,绝缘子结合蛋白,CTCF基因编码的转录因子,与绝缘子的活性相关,CTCF与靶顺式元件的结合可阻断增强子和启动子的相互作用,从而将增强子的活性限制在一定的功能区域。)
TADs的存在也提示了基因和远端调控元件之间可能发生的loop互作会存在限制,loop互作局限于位于相同TAD内的元件。事实上,小鼠基因组中的初步分析表明增强子-启动子相互作用在TAD内特别频繁。也就是,TAD在调控基因表达方面具有主要作用,将基因限制于某些特定的远端调控元件。对X染色体失活中心的TAD进行分析表明,相同TAD内的基因倾向于在细胞分化期间协调表达,可能是因为它们共享相同的一组基因调控元件。 TADs的存在可以为长期观察到一种现象提供染色质结构层面的解释,这种现象即相邻基因通常在多种细胞类型中表达相关。
七、补充内容:基因组隔间(Genome compartments)
哺乳动物基因组的染色体内和染色体间相互作用图揭示了一种相互作用模式,可以近似分为两个隔间(A和B,或称区室/隔室),它们沿着染色体交替,并且具有约5Mb的特征尺寸。A隔间优先与整个基因组中的其它A隔间相互作用。 同样,B隔间与其它B隔间相关联。隔间信号可以通过互作图的特征向量扩展来量化。 A / B室信号不是简单的双相(仅代表两种状态),而是连续的,并且与转录活性指标,如DNA可及性、基因密度、复制时间、GC含量和几个组蛋白标记相关。A-隔间主要是常染色质-转录活跃区域,B-隔间则主要是聚集在一起的异染色质(转录失活区域)。
Compartment的发现:Lieberman-Aiden 等(2009)在研究人染色质互作时发现,在分辨率为1 Mb时,得到的染色质相互作用矩阵图中,由于染色质不同区间互作强度不同产生了明显的“格子图案”模型(plaid pattern),见下图,从而将染色质大致分成2部分,A 隔间和B隔间。
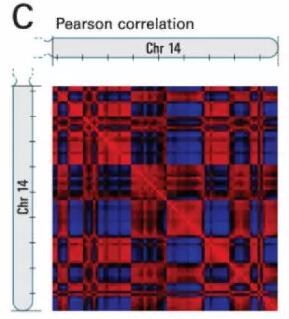
图片引自Comprehensive mapping of long-range interactions reveals folding principles of the human genome[J]. science, 2009, 326(5950): 289-293,互作矩阵中颜色表示每对1Mb基因座染色质间互作相关性系数(红色:1,蓝色:-1)。
TAD不同于较大的A和B隔间。:1)对胚胎干细胞、脑组织和成纤维细胞的分析表明,大部分的TAD在不同组织间保持不变,而A隔间和B隔间是活性和非活性染色质的组织特异性结构域,其与细胞类型特异性基因表达模式相关;2)A隔间和B隔间通常为几Mb,并沿着染色体形成活性区域和非活性区域交替模式,相比之下,TAD较小(中值约为400-500Kb),可以是活性的或无活性的,并且相邻的TAD不一定具有相反的染色质状态。 因此,TADs似乎是染色体的硬件特征,并且一组相邻TADs形成A隔室和B隔室。
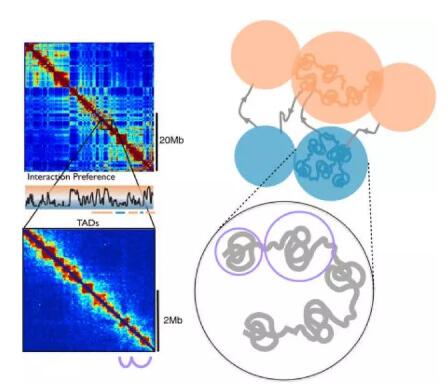
未完待续:后面的部分着重阐述构建染色质的3D模型构建方法,即前面引言所述的另外2种方法:距离约束建模(restraint-based modeling)方法和聚合体(polymer)建模方法,由于篇幅所限,暂不列在本次解读中。
如果您对该技术感兴趣,欢迎点击下方按钮联系我们,我们将免费为您设计文章思路研究方案。
参考文献:
Dekker J, Marti-Renom M A, Mirny L A. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data[J]. Nature Reviews Genetics, 2013, 14(6): 390.









 京公网安备 11011302003368号
京公网安备 11011302003368号