
棉花为世界纺织品提供着优质纤维,在4种棉花栽培品种中,陆地棉(Gossypium hirsutum)以其高产量,纤维品质适中,强适应性的特点在生产中占世界棉花的95%,备受人们关注。自2016年以来,中国农业科学院棉花研究所与北京百迈客合作的棉花遗传图谱文章相继发表于Frontiers in Plant Science、BMC Plant Biology、Crop Science[1,2,3],分别对陆地棉的黄萎病抗性,纤维强度,棉铃重性状进行了QTL定位。近期,华中农业大学张献龙教授,林忠旭教授和百迈客合作的基于SLAF-seq测序技术,利用陆地棉和毛棉的种间杂交渗入系定位影响棉花产量和纤维品质的QTL发表于International Journal of Molecular Sciences [4]上,具体做了什么呢?图谱君给大家详细解读。
PS:文末同样有中标的基金标书哦~

1.材料与方法
1.1群体构建
父母本:Gossypium hirsutum acc.4105(轮回亲本)× G. tomentosum (供体亲本)
群 体:107个IL群体(图1为群体构建过程)
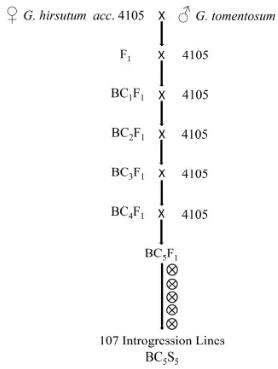
图1 群体构建流程
1.2表型鉴定
两年三点试验,分别对IL群体的107个个体及其父母本的8个产量性状,5个纤维品质农艺性状考察。
8个产量性状包括:铃重(BW),棉重(LW),皮棉百分率(LP),百粒重(HSW),株高(PH),铃数(BN),果实分枝数(FBN)和种子指数(SI);
5个纤维品质性状包括:马克隆值(MIC),纤维长度(FL),纤维整齐度(FU),纤维强度(FS)和纤维伸长率(FE)。
1.3 SNP Calling
应用Illumina High-seq 2500测序平台。
在轮回亲本中,获得595419个SLAF标签;供体亲本中,获得359255个SLAF标签;IL群体中,获得258697个SLAF标签,共得到3157个SNP位点。
1.4 QTL定位
利用QTL IciMapping 4.1软件进行QTL定位。
2结果分析
2.1表型考察
作者分别对IL群体的107个个体及其父母本的8个产量、5个纤维品质农艺性状两年三点表型考察,所关注的性状在IL群体个体间存在显著变化(p<0.01水平),其中棉铃数的变异系数(CV)值为24.91-25.43,显著高于其他性状,纤维整齐度的CV为1.27-1.52,显著低于其他性状。IL群体13种性状表型值均表现出正态分布,表明每个性状都受多基因控制。
2.2 SLAF-Seq及SNP开发
对供体亲本、轮回亲本及107个渗透系群体SLAF测序数据分析,Q30达到91%,GC含量为40%,表明得到较高质量的测序数据。轮回亲本中,共获得595419个SLAF标签,每个标签的平均测序深度为29.73×,供体亲本中获得359255个SLAF标签,平均测序深度为21.12×,ILs群体中,获得258697个SLAF标签,每个SLAF标签的平均测序深度为8×。基于测序所得的SLAF标签,共得到了3157个SNP多态性标记,其中1816个位于A基因组,1341个位于D基因组。每个染色体上的SNP标记数量分布范围从24-535不等(图2)。
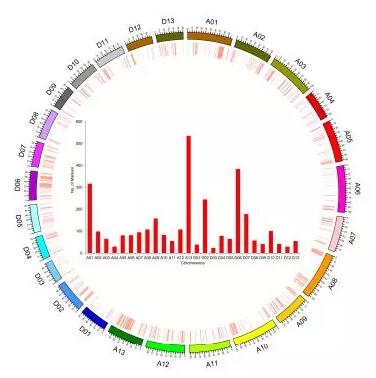
图2 不同染色体上的marker数
2.3 基因组组成及ILs多样性分析
使用Microsoft Excel计算基因组中渗入片段的覆盖度和百分比,结果表明,渗入片段占四倍体棉花基因组的35.13%,A基因组和D基因组分别为35.68%和34.59%。最大的渗入片段位于A13染色体,大小为62Mb,且A13染色体上渗入片段基因组覆盖率最高,为77.54%。最小的渗入片段位于D09染色体,大小为9Mb,D11染色体上渗入片段基因组覆盖率最低,为19.67%。
2.4 影响纤维品质及产量的QTL定位
在20条染色体上共检测到74个与纤维品质和产量相关的QTL。其中,30个QTL与5个纤维品质性状相关,44个QTL与8个产量性状相关。其中47个QTLs(63.51%)来自于A基因组,27个QTL(36.49%)来自D基因组。共发现69个SNP标记与74个QTL相关,其中29个标记与纤维品质性状相关,40个标记与产量相关。13个性状的的PVE介乎2.02%-30.15%之间,平均为10.22%。
2.5 QTL 热点分析
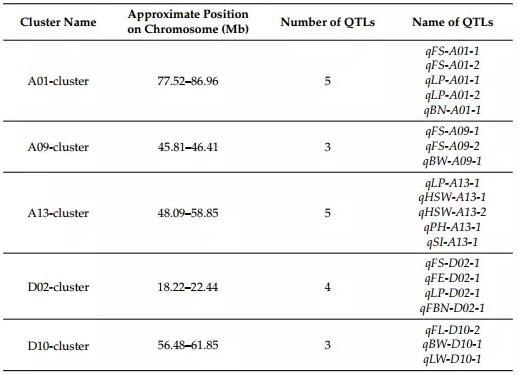
作者以10 Mb(〜20 cM)物理区域包含两个或多个QTL定为QTL簇标准。在5条染色体上检测到5个QTL簇,每个簇至少有3个QTL(表1)。其中3个位于A基因组,2个位于D基因组中。鉴定出含有影响纤维品质和产量性状的多个QTL的基因组区域,有利于后期分子改良。
表1 影响产量及纤维品质的QTL簇位点分析

参考文献:
[1] Palanga K, Jamshed M, et al. Quantitative Trait Locus Mapping for Verticillium wilt Resistance in an Upland Cotton Recombinant Inbred Line Using SNP-Based High Density Genetic Map [J], Frontiers in Plant Science, 2017.
[2] Zhen Z, Qun G, et al. Construction of a High-Density Genetic Map and Its Application to QTL Identifcation for Fiber Strength in Upland Cotton [J], Crop Science, 2017
[3] Zhen Z, Hai H, et al. Construction of a high-density genetic map by specific locus amplified fragment sequencing (SLAF-seq) and its application to Quantitative Trait Loci (QTL) analysis for boll weight in upland cotton (Gossypium hirsutum.)[J], BMC Plant Biology, 2016
[4] Ayaz A, Chao S, et al. QTL Mapping for Fiber Quality and Yield Traits Based on Introgression Lines Derived from Gossypium hirsutum × G. tomentosum[J], International Journal of Molecular Sciences, 2018
遗传群体事业部 胡思帆丨文案
许语辉 | 审核
图片来自网络,侵删









 京公网安备 11011302003368号
京公网安备 11011302003368号