大豆作为重要的农作物,在我国有着6000-9000年的驯化史,其驯化方向主要与产量、品质和抗病等性状相关。东北农业大学的李文滨教授及其团队和北京百迈客生物科技有限公司合作,在大豆起源、抗菌核病、抗胞囊线虫、种子蛋白及油脂含量方面取得重要进展,其研究成果相继发表在New Phytologist、The Plant Journal、BMC Genomics和Genomics,累计影响因子23+。下面我们就看看作者是如何对数据进行深入挖掘,做到低投入、高产出的。
1.英文题目:Domestication footprints anchor genomic regions of agronomic importance in soybeans
中文题目:512株大豆SLAF测序揭示大豆驯化史和全基因组关联分析
发表杂志:New Phytologist,2015
影响因子:IF= 7.330
1.材料与方法
(1)材料:512份大豆材料,470份国内材料(404份驯化材料、36份半驯化材料和72份野生大豆)+42份国外材料。
(2)方法:SLAF标签数为59494条,平均测序深度为6.14×。
2.研究结果
(1)采用百迈客研发的SLAF-seq技术对512份大豆核心种质进行高通量测序分析,共获得了64141个高质量SNP。
(2)通过基因渗透和PCA分析推出半驯化品种是一种进化上的中间产物,而不是驯化品种和野生大豆杂交产生的,此外,三群体检测的结果也进一步论证了这一点,同时,论证出栽培大豆是从中部黄淮海地区向北向南驯化,进而将中国驯化大豆起源√确的定位到中国中部黄淮海区域。
(3)基于SNP分型结果,定位到与15个性状相关的43个区域,如与茸毛和种皮颜色相关的基因被定位在Gm06染色体,与叶形相关的基因被定位在Gm20染色体。在与10个质量性状相关的25个QTNs中,有3个QTNs位于之前已经报道过的QTLs或基因区域。
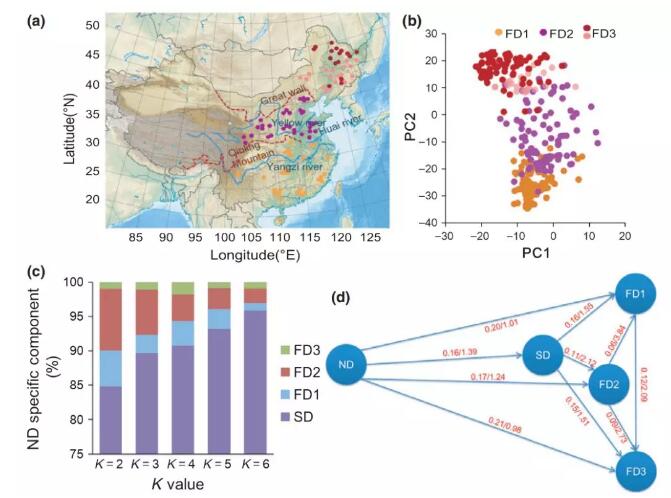
Fig1: 栽培大豆的地理分布和PC分析图
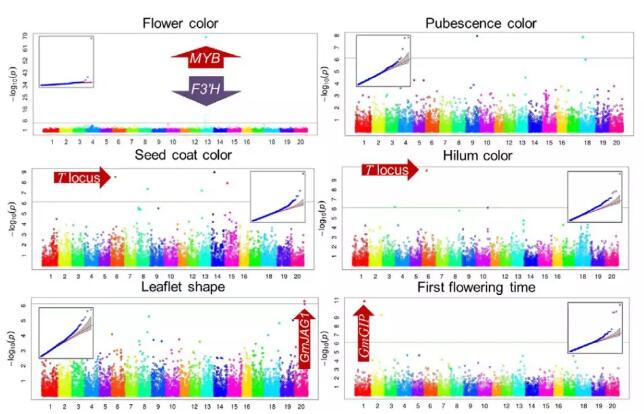
Fig2:重要农艺性状的群基因组关联分析图
2.英文题目:Loci and candidate gene identification for resistance
to Sclerotinia sclerotiorum in soybean (Glycine max L. Merr.) via associate
中文题目:330株大豆SLAF-GWAS及遗传图谱联合定位抗菌核病基因
发表杂志:The Plant Journal,2015
影响因子:IF= 5.901
1.材料与方法
(1)材料:330份大豆材料(105份地方材料、201份骨干材料和24份国外材料)进行GWAS分析;以Maple Arrow(抗病)和Hefeng 25(感病)为亲本构建128份重组自交系,通过SSR标记构建遗传图谱。
(2)方法:SLAF标签数为57418条,平均测序深度为6.02×;利用78个SSR标记构建遗传图谱。
2.研究结果
(1)茎脂溶性色素含量是大豆抗菌核病的重要指标。通过测定OD518值发现,亲本Maple Arrow和Hefeng 25的OD518值分别为0.1 与0.0001,128份RIL群体和330份种质的OD518值成正态分布。
(2)在前人研究基础上,利用Gm01, Gm08, Gm11和Gm13上的78个SSR构建遗传图谱,在Gm13上定位到主效QTL位点Oswm13-1。
(3)基于获得的25179个SNP,采用Naive、GLM 和CMLM 三种模型进行关联分析,在Gm13上定位到3个热点标记,通过比较发现GWAS和遗传图QTL定位结果是重叠的。进一步对候选基因进行功能注释,有4个基因与植物病害应答机制或色素合成有关。
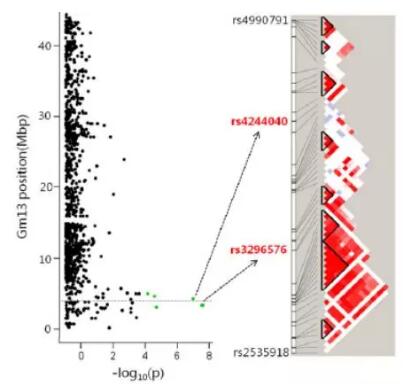
Fig.1 大豆抗菌核病全基因组关联分析
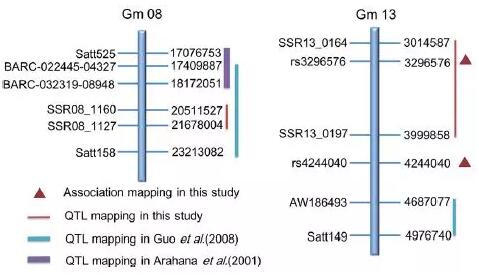
Fig.2 QTL定位结果
3.英文题目:Genetic charactertics of soybeanresistance to HG type 0 and HG type1.2.3.5.7 of the cyst nematode analyzed bygenome-wid association mapping
中文题目:440株大豆SLAF-GWAS定位胞囊线虫抗性基因
发表杂志:BMC Genomics,2015
影响因子:IF= 3.729
1.材料与方法
(1)材料:440份大豆材料,402份国内材料和38份国外材料
(2)方法:SLAF标签数为57 418条,平均测序深度为6.02×;利用78个SSR标记构建遗传图谱。
2.研究结果
(1)基于SLAF-seq技术对440份大豆核心种质进行高通量测序分析, 共获得了36,976个高质量SNP。
(2)通过全基因组关联分析找到19个与胞囊线虫抗性相关的位点,其中8个位点与之前报道过的QTL位点重合,8个位点与已经报道的抗胞囊线虫基因Rhg4及Rhg1连锁,并鉴定出3个新的QTL位点。通过基因注释获得到与抗病,细胞色素P450,蛋白激酶等相关的功能基因。
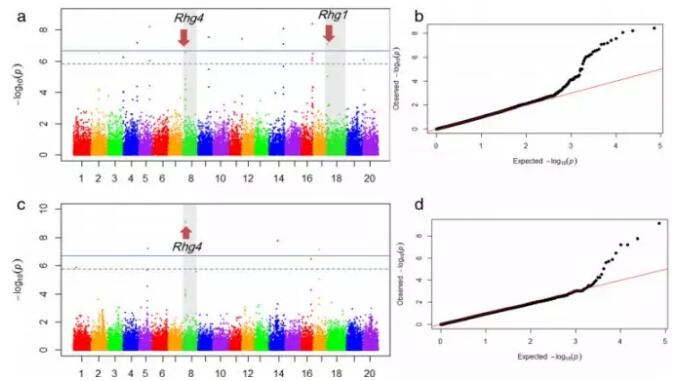
Fig.1 全基因组关联分析定位大豆胞囊线虫QTL位点
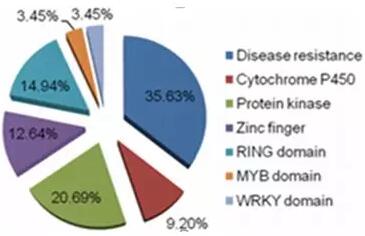
Fig.2 基因功能注释图
4.英文题目:Loci and candidate gene conferring resistance to soybean cyst nematode HG type 2.5.7
中文题目:200株大豆SLAF-GWAS定位胞囊线虫抗性位点及基因
发表杂志:BMC Genomics,2017
影响因子:IF= 3.729
1.材料与方法
(1)材料:200份大豆材料,179份国内材料(123份骨干材料和56份地方材料)和21份国外材料
(2)方法:SLAF标签数约为58,000条,平均测序深度为6.14×。
2.研究结果
(1)基于SLAF-seq技术对200份大豆核心种质进行高通量测序分析, 共获得了33,194个高质量SNP。
(2)采用GLM、CMLM 和ECMLM 三种模型进行关联分析,共获得与胞囊线虫抗性相关的13个SNP位点,9个SNP位点位于已知QTL区域附近,4个SNP位点是新发现的抗病位点,通过功能注释共获得30个抗病候选基因,主要与植物防御、木质素生物合成、酚类化合物代谢等相关。
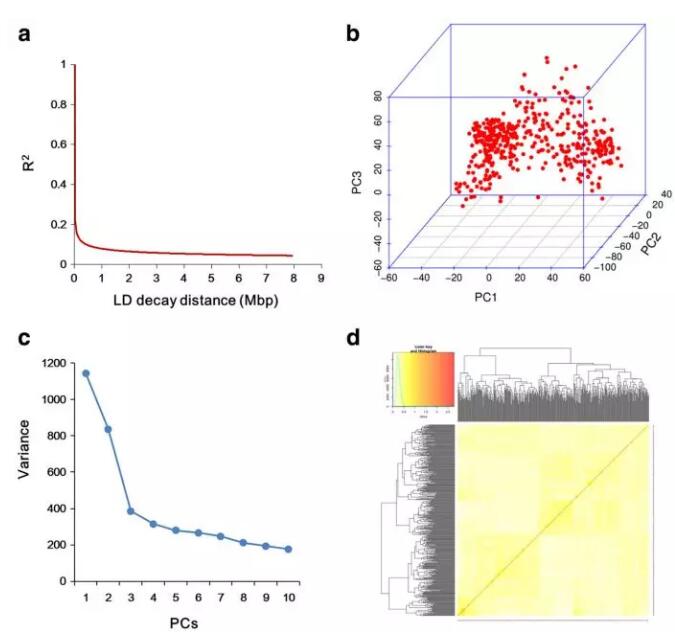
Fig.1 群体结构
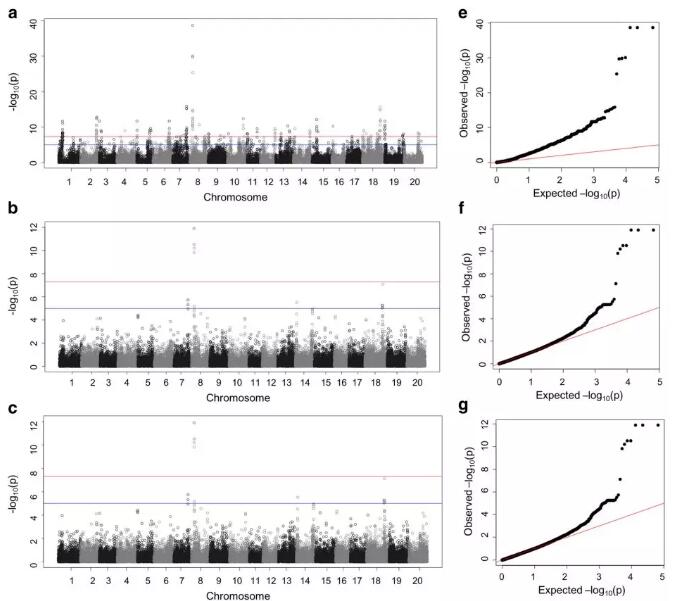
Fig.2 全基因组关联分析定位大豆胞囊线虫QTL位点
5.英文题目:Genome-wide association mapping for seed protein and oil contents using a large panel of soybean accessions
中文题目:185株大豆SLAF-GWAS定位种子蛋白及油脂含量
发表杂志:Genomics,2018
影响因子:IF= 2.801
1.材料与方法
(1)材料:185份大豆材料(骨干材料、地方材料和国外材料)
(2)方法:SLAF标签数大于50,000条。
2.研究结果
(1)基于SLAF-seq技术对185份大豆核心种质进行高通量测序分析, 共获得了12,072 个高质量SNP。
(2)采用GLM、CMLM 和ECMLM 三种模型进行关联分析,共获得与种子蛋白及油脂含量相关的31个SNP位点,在2015年和2016年分别有6和9个SNP位点与种子油脂含量关联,其中2个SNP位点在两年的表型数据中均能关联到,位于Chr 07上的rs15774585和rs15783346,在2015年和2016年分别有14和7个SNP位点与种子蛋白含量关联,其中3个SNP位点在两年的表型数据中均能关联到,位于 Chr 01上的rs53140888,Chr 13上的rs19485676和Chr 20上的rs24787338,通过功能注释共获得199个候选基因,161个基因主要与CHO代谢,脂质代谢和氨基酸代谢等相关。
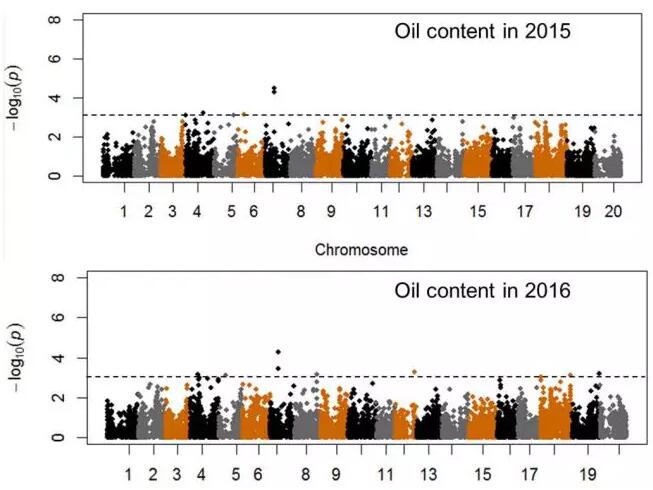
Fig.1 全基因组关联分析定位大豆油脂及蛋白QTL位点
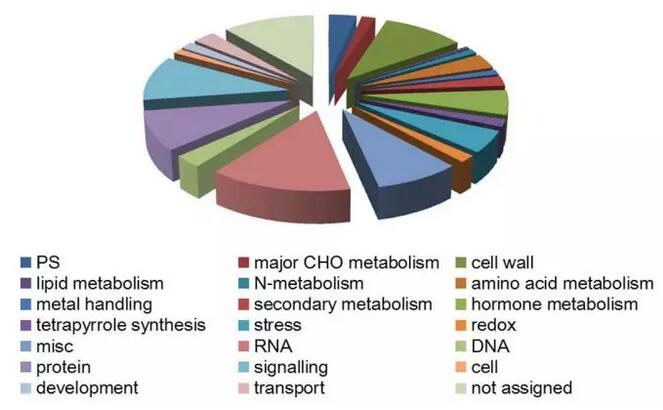
Fig.2 基因功能注释图

基因组事业部 田韦韦 | 文案
张学雯 | 审核
图片来自网络,侵删








 京公网安备 11011302003368号
京公网安备 11011302003368号