本篇文章研究的物种为高杂合度的南美白对虾,采用Illumina Hiseq 2500,PE100测序平台,SLAF-seq简化基因组测序技术,对两个亲本及149个F1子代个体进行测序分析。
研究结果
本研究所用的群体类型是F1,149个个体,利用SLAF-seq技术对样品进行高通量测序开发分子标记。共获得456,620,260个reads,开发了114,829个SLAF标签,其中多态性标记25,140个,成功编码的高质量标记6359个,采用拟测交的原理构建了包含44个连锁群的遗传图谱,总图距为4,271.43 cM,平均图距为0.7cM,其中雄性图包含4201个标记,雌性图包含4396个标记,中性图包含6146个标记,同时对构建的图谱进行了独创性的图谱评价。
白对虾部分连锁群
结合白对虾生长性状的数据分析,定位到11个与体长紧密相关的QTL位点,7个与体重相关的QTL位点,分布于不同连锁群上,其中只有Marker 7605与两种性状同时显著相关,对于后续的分子标记辅助育种具有很好的利用价值。
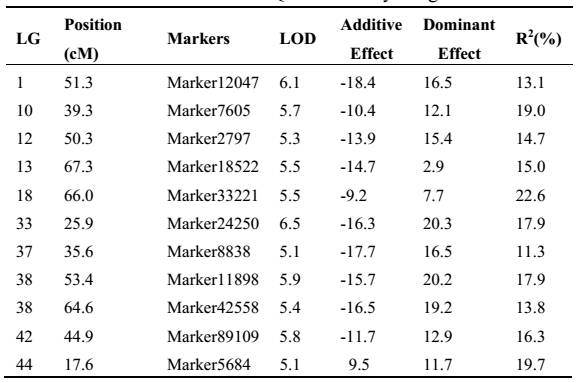
体长相关的QTL
利用区间作图法,定位到体重和体长QTL共11个,共可解释表型变异为38.56%。其中位于连锁群33标记24250位置的QTL解释表型变异达到17.9%,LOD值为6.5,为一主效QTL。
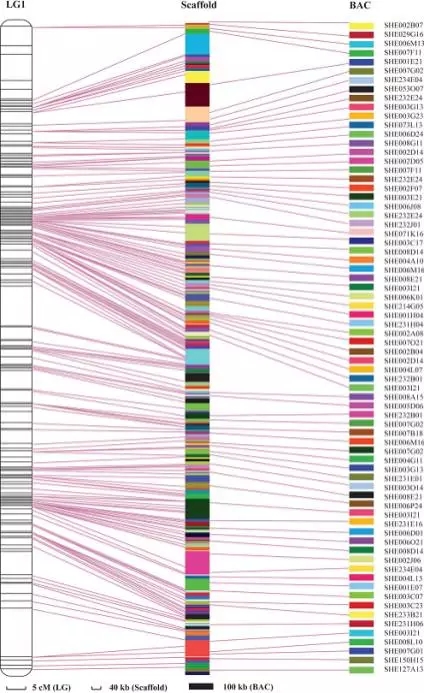
遗传图、Scaffold、BAC共线性比较
白对虾是一个高杂合物种,其基因组含有80%的重复序列,其基因组组装是一个很大的难题。本研究通过对遗传图谱、白对虾初步组装的Scaffold、BAC测序的序列进行共线性比较,最终使得5922个标记锚定到5885个Scaffold上,有利于后续白对虾的基因组组装。
参考文献
Genome survey and high-density genetic map construction provide genomic and genetic resources for the Pacific White Shrimp Litopenaeus vannamei .







 京公网安备 11011302003368号
京公网安备 11011302003368号