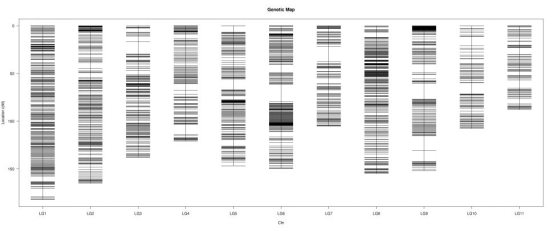
遗传图谱
经典的遗传学研究方法
遗传图谱是依据染色体交换与重组,以多态性的遗传标记为路标,以标记间的重组率为“图距”,
确定不同多态性标记位点在每条连锁群上排列的顺序和遗传距离的线性连锁图谱
应用领域

针对性的个性化方案
从物种个体取样到候选基因挖掘,我们将为您提供一站式个性化解决方案,让分析过程快速、科学、清晰!
-
物种取样
-
建库测序
-
标记分型
-
图谱构建
分析内容
测序数据下机后,每一个分析过程我们都严格把控,针对不同的测序方式,从数据质控到遗传图谱的构建,有专门的的流程进行分析。
| 全基因组重测序 |
| 测序数据质控 |
| 与参考基因组比对 |
| 变异检测及注释 |
| 多态标记开发 |
| 遗传图谱构建及评估 |
| 简化基因组测序(SLAF) |
| 测序数据质控 |
| 与参考基因组比对(有参) |
| 标签聚类比对(无参) |
| SNP变异检测及注释 |
| 多态标记开发 |
| 遗传图谱构建及评估 |
SLAF简化遗传图谱
SLAF-seq技术是百迈客公司自主研发的一套基于酶切的简化基因组测序策略,
不受参考基因组限制,能够有效地降低物种基因组复杂度,
SLAF标签构建遗传图谱具有上图标记数量多、图谱质量高、周期短、数据利用率高等的优点。
重测序遗传图谱
对已有参考基因组序列的物种进行群体全基因组重测序,利用高性能计算平台和生物信息学方法,
检测单核苷酸多态性位点(SNP),并计算多态性标记间的遗传连锁距离,
绘制高密度的遗传图谱, 在进行QTL定位中,可直接获取定位区间内的序列信息,加快功能基因的鉴定和验证的进度
百迈客优势

研发简化基因组测序技术SLAF-seq
专门针对高通量测序开发的HighMap作图软件
丰富的项目经验,已完成百余个物种超过近千张高密度遗传图谱
截止目前已服务客户发表SCI文章200+篇,累计影响因子700+
成功案例
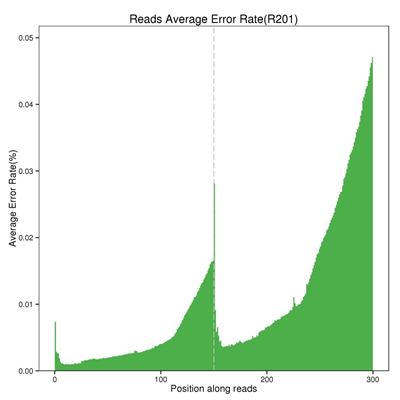
测序数据统计与评估
高质量的测序数据是进行后续分析的重要基础,在获得测序的Raw data后,我们将会进行测序数据产出统计、碱基测序质量分布、碱基类型分布等多种测序评估方式,保证测序数据的准确性。
| #Chr | Pos | Ref | R01 | R02 | … |
| Chr1 | 113342 | A | G | A | … |
| Chr1 | 163871 | A | G | A | … |
| Chr1 | 232230 | A | A | G | … |
| Chr1 | 232330 | C | C | A | … |
| Chr1 | 232546 | T | T | C | … |
群体遗传变异检测
SNP(Single Nucleotide Polymorphism)及InDel(Insertion-Deletion)是基因组上最为常见的遗传标记,具有数量多、多态性丰富的特点,SNP和InDel的检测主要通过GATK和samtools软件工具包实现。变异位点注释使用SnpEff软件。
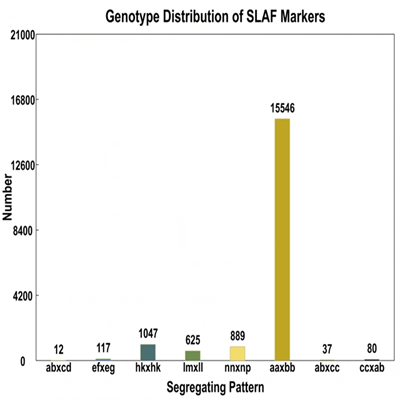
多态标记开发
在变异位点检测的基础上,开发亲本间的多态性标记,并对子代中这些多态性标记进行基因型分型。
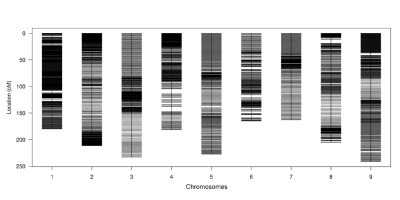
遗传图谱构建
为保证遗传图谱质量,对子代中多态性标记进行严格的筛选过滤,包括亲本深度过滤、异常位点过滤、标记完整度过滤及偏分离标记过滤等,获得高质量标记。利用百迈客自主研发的Highmap-map软件进行高密度遗传图谱的绘制。
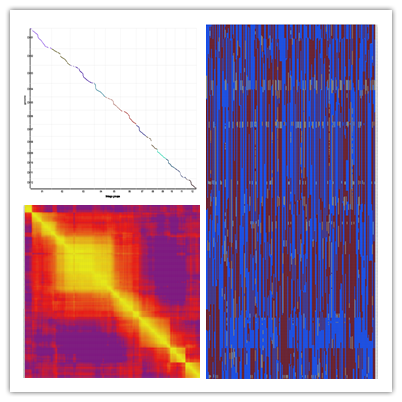
遗传图谱评估
为了保证遗传图谱的质量,百迈客采用了目前最为严格的图谱评价体系,包括上图标记完整度统计、单体来源评估、连锁关系评估、遗传图谱共线性分析等。
根据亲本杂合、纯合情况选择分离群体类型:
(一)亲本杂合的物种,如林木、水产、果树等异花授粉、虫媒、风媒授粉、雌雄异体物种,一般采用F1。
(二)亲本纯合物种,如水稻、豆类等自花授粉,分离群体可用F2、BC、RIL、DH、巢式群体等。
两个亲本推10-20×,子代测序深度看物种基因组组装程度,如果组装结果不好的话,大部分片段都比较短,可能最后会影响遗传图谱的结果,推荐测序深度3X以上,如果基因组装到染色体水平或者N50达到Mb级别测1×就可以了。
需要,9311基因组虽然出来,但是9311有很多不同类型的突变,比如有的有芒,有的无芒,建议老师测,但是测序深度可以稍微降低。
大部分QTL定位方法只是要求表型数据中的随机误差项服从正态分布,并没有要求表型数据满足正态分布。数量性状只有在多基因假说下才真正符合正态分布,表型数据的非正态性并不影响QTL定位。
包含偏分离不显著的标记,显著偏分离的标记会被过滤掉。偏分离是自然界非常普遍存在的现象,并被认为是生物进化的动力之一。产生偏分离的原因主要有两个方面:配子选择和合子选择,其中配子选择主要包括花粉致死、花粉管竞争和选择性受精。除了上述原因以外,环境因素、非同源重组、基因转换、转座因子、转基因沉默、体外孤雄生殖过程中的选择压、非整倍体或不稳定易位造成的染色体不稳定等都有可能是导致偏分离。
通过共线性分析,将遗传图谱上的标记与初步组装得到的scaffold进行比对,统计不同scaffold上的标记数目,得到不同连锁群上被锚定的scaffold信息,一个scaffold上如果锚定两个以上的标记,该scaffold的位置和方向均可以被定位到染色体水平。只有1个标记则只能定位到位置,方向不能确定。如果没有锚定到标记,则该scaffold仍然不能被定位到染色体水平,说明该scaffold多态性可能较差,需要构建更加精细的遗传图谱。
判断标记是否偏分离是采用的卡方检验,卡方检验的显著性P值越小,越证明该标记偏分离严重,因此P值小于0.001的标记要少于P值小于0.01的标记的数量。在进行过滤的时候如果按照P值<0.001过滤,剩余的标记会更多,因此是比较宽松的过滤标准,反之,按照P值<0.01过滤,会过滤掉更多的标记,是更为严格的过滤标准。
取F2的叶片,F2:3家系的表型平均值作为F2的表型,群体要随机取样(F2:3家系是指F2代群体中,每个F2代植株自交又产生的各自的F3群体组成的群体,这个F3是与上一代F2所对应的,所以也称为F2:3家系,他们是不能混在一起的,混在一起就叫F3了,而不是F2:3。这种群体主要是用于初步定位的,对于单显性基因控制的质量性状定位效果很好。)
无需建立BAC库,亲本重测序可以获得QTL区域的基因序列,进行基因预测和后续标记开发转基因验证等工作。
1)表达数量性状基因座(expression Quantitative Trait Loci,eQTL)是指的是染色体上一些能特定调控mRNA和蛋白质表达水平的区域,其mRNA/蛋白质的表达水平量与数量性状成比例关系,将各基因型的表达数据作为一个数量性状,利用传统的QTL分析方法进行分析。eQTL可分为顺式作用eQTL和反式作用eQTL,顺式作用eQTL就是某个基因的eQTL定位到该基因所在的基因组区域,表明可能是该基因本身的差别引起的mRNA水平变化;反式作用eQTL是指某个基因的eQTL定位到其他基因组区域,表明其他基因的差别控制该基因mRNA水平的差异。
2)基于转录组测序的关联分析是从SNP关联分析与GEM关联分析两个水平实现的,开发的SNP位于基因编码区域,SNP与基因(或Unigene)所属关系是已知,加上基因表达水平的关联分析就能够实现功能基因的精细定位。
主要有四款:R-qtl、MapQTL、WinQTLcart和ICiMapping。
这是由于标记的子代分型依赖于亲本的分型造成的。遗传图中每一个标记的子代分型首先要根据亲本的分型来进行分型判断,因此亲本分型的准确性就很重要,是依靠高深度的测序来确保亲本分型的准确性,因此在测序的时候要比子代的测得多。
- Mol Plant丨转录组学和群体研究联合助力高粱功能基因组学研究和遗传育种2025年3月7日,中国科学院遗传与发育生物学研究所谢旗研究员团队联合中国农业大学于菲菲教授团队在Molecu […]阅读更多






 京公网安备 11011302003368号
京公网安备 11011302003368号