医学Nanopore全基因组重测序
产品介绍
重测序是对已知基因组序列的物种进行不同个体的基因组测序,并进一步对个体或群体进行差异性分析。
Nanopore测序借助单个分子通过纳米孔时引起孔两侧电位差来实现信号检测,纳米孔的直径仅允许单个核苷酸聚合物通过,而ATCG四种碱基的带电性质不同,因此通过电信号差异特征即可检测出通过纳米孔的碱基类型,从而实现测序。

技术优势
实验流程
实验流程按照 Oxford Nanopore Technologies(ONT) 公司提供的标准 protocol 执行,包括样品质量检测、文库构建、文库质量检测和文库测序等流程。
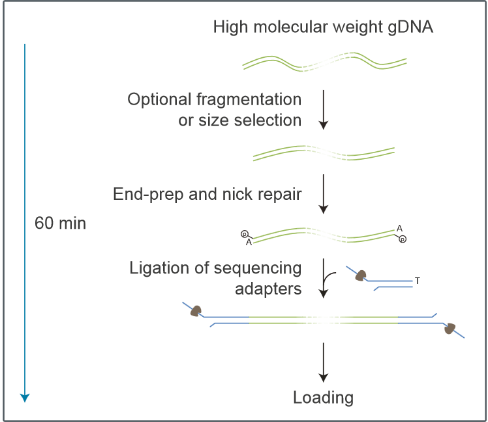
重测序分析流程
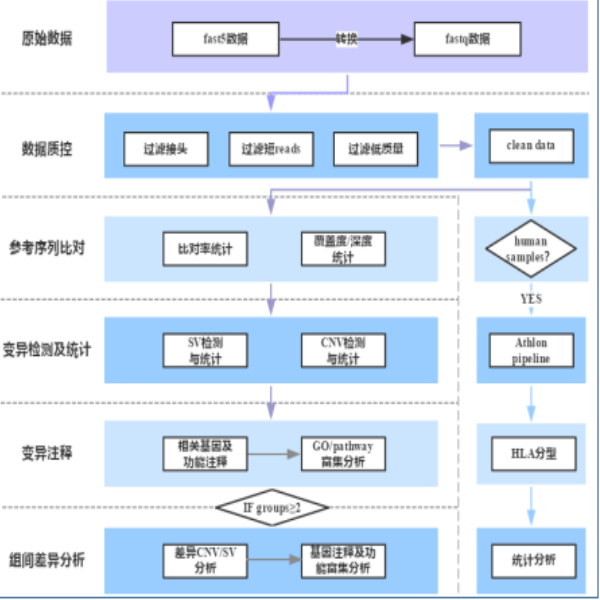
结果展示
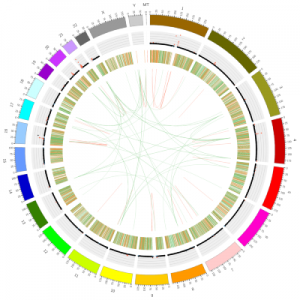
结构变异圈图
根据检测得到的变异信息整合成 Circos 图,对基因组数据进行可视化展示。
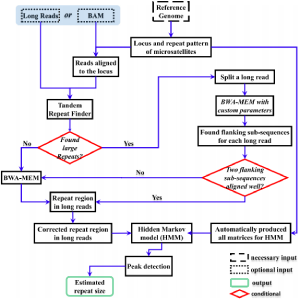
Repeat分析
对测序数据进行串联重复序列分析,可以检测重复元件的核心序列和重复次数。
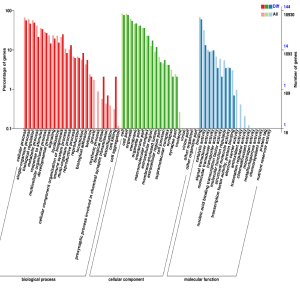
差异CNV/SV的基因注释
对识别到的差异 CNV/SV 进行基因注释,并对这些基因进行功能注释及富集分析,GO注释分类统计图,直观的反映出在生物过程(biological process)、细胞组分(cellular component)和分子功能(molecular function),所有基因和差异基因注释GO term的个数分布。
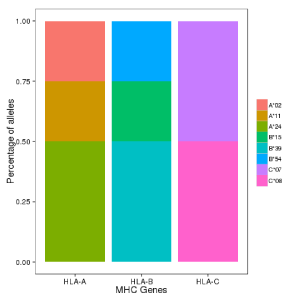
HLA分型
若样本为人,则会有HLA分型分析,可展示样本在不同分辨率下的等位基因的多样性。横坐标代表不同的 HLA 基因,纵坐标代表每个等位出现的频率。如果基因中颜色的种类越多说明该基因在所研究的群体中的其多样性越高
常见问题
片筛和非片筛文库的区别是什么?
片筛即片段筛选,如果片筛文库则会选择长的DNA片段,因此需要的DNA量要多;非片筛文库即不进行片段筛选的打断文库,即将DNA打断成固定长度。
片筛文库得到的序列相对更长,适用于基因组组装。而非片筛文库得到的read相对较短,适用于重测序或甲基化检测。
重测序一般要测多少数据量?
原则上讲数据量的越多,检测到的结构变异越多越准。由于经费限制推荐至少测15X的数据量,如果是肿瘤等特殊组织建议测15X以上的数据量。具体原因可参考下述链接:








 京公网安备 11011302003368号
京公网安备 11011302003368号