BSA关联定位分析
高效快速基因定位策略
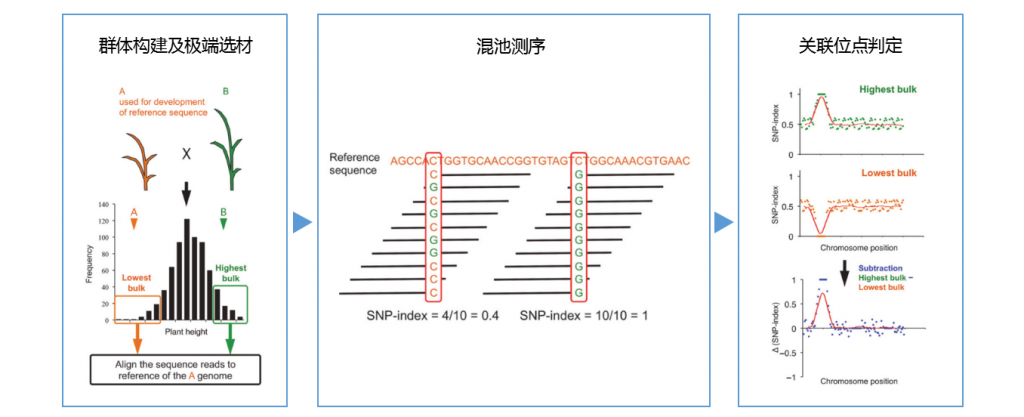
BSA分析是将分离群体中表现出极端性状的个体混合起来构建两个混池,通过分析两个混池间差异序列,快速定位与目的基因紧密连锁分子标记的分析方法,该方法广泛应用于动植物基因定位中。
个性化分析方案
BSA分析以其快速、准确、性价比高的分析特点在许多物种的几十个性状上得到广泛应用
针对特定的群体和性状,我们将会推荐合理的分析方案
-
物种取样
-
建库测序
-
标记开发
-
关联分析
分析内容
根据物种基因组大小、复杂度和研究目的,可以选择不同的分析策略,包括重测序-BSA及其突变体分析形式Mutmap。
测序数据质控;与参考基因组比对;SNP、InDel检测和注释;关联分析;关联区域注释
技术优势
百迈客成功完成了近1000个物种的10000多个性状的定位工作
项目经验及其丰富,目前已发表文章80+,累计影响影子300+
可根据项目需要选择最佳分析方案,保障结果精准
选材要求及测序方案
| 取样要求 | 测序方案 | 项目周期 |
| 重测序-BSA需要有参考基因组的物种 | Illumina测序平台,PE150测序策略,保证Q30达到80% | 45天 |
| 具有重要农艺性状个体构建的遗传群体(F2、RIL、DH、BC等)) | 推荐混池中每个子代测序深度≥1×,每个亲本≥20× | |
| 子代混池规模:质量性状不低于30+30,数量性状推荐选取群体规模的5%-10%,同时不低于30+30的个体数 |
测序数据统计与评估
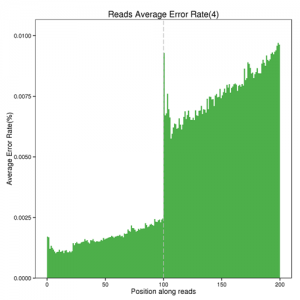
高质量的测序数据是进行后续分析的重要基础,在获得测序的Raw data后,我们将会进行测序数据产出统计、碱基测序质量分布、碱基类型分布等多种测序评估方式,保证测序数据的准确性。
遗传变异检测
SNP(Single Nucleotide Polymorphism)及InDel(Insertion-Deletion)是基因组上最为常见的遗传标记,具有数量多、多态性丰富的特点,SNP和InDel的检测主要通过GATK和samtools软件工具包实现,变异位点注释使用SnpEff软件。
关联分析
根据亲本和混池的SNP及Indel标记,利用SNP-Index和ED分析方法同时进行标记与性状的关联分析,获得与目标性状相关的基因。
关联区域注释
应用BLAST软件对候选区间内的编码基因进行多个数据库(NR、Swiss-Prot、GO、KEGG、COG)的深度注释,通过详细的注释,进一步缩小关联区域,快速筛选候选基因。
成功案例
对于数量性状定位的混池,通常建议混池规模为群体大小的5%-10%。不同的测序策略,混池规模会有略微的差别,重测序BSA及转录组BSA最低要求30个以上,SLAF-BSA最低要求50个以上。文献报道(300样/池&160样/池相比)混池规模越大,定位的效果会越好。
常测序数据量越大,标记的准确性会越高,但成本也会越高,综合考虑,在这里我们给大家提供BSA项目的最低数据量。对于SLAF-BSA,通常我们建议子代混池至少达到每个个体1×的数据量,比如混池规模为30+30,那么每个混池测至少30×;对于重测序BSA,混池规模在50以下,子代1×的数据是可以的,但是像基因组比较大的物种(如玉米),若混池规模为100+100这样的情况,每个混池测50×也是没有问题的。
能做。现在我们的BSA关联分析是基于SNP,因为SNP在基因组上的密度要远远高于Indel以及结构变异,这会使我们定位更加精细。如果某一个性状是由Indel、结构变异、导入系所引起的,那么它通常是与某些SNP连锁发生的,通过SNP我们会找到候选区域,再在候选区域内寻找这些Indel或结构变异或导入系,来寻求决定该性状的Indel或结构变异或导入系。
可以,对于没有亲本的混池,我们会采取ED的方法进行关联分析。
有影响。从关联分析的方法上,有亲本的BSA我们会有两种关联分析方法,SNP-Index和ED,最后的定位结果是两种方法的交集,无亲本的只能用ED的方法进行关联分析。有亲本的作用是排除亲本非多态性标记对定位的影响,举例说明一下,有一个标记在混池间是有多态性的,但在亲本上是没有多态性,如果有亲本,我们的定位结果是不会有这样的标记,但是没有亲本,定位结果中就会有这样的标记,定位的区域会变大,假阳性较多。
不可以,两个不同群体所采用的阈值没法计算。
推荐至少做两个亲本的重测序,这样我们会提供定位区域内非同义突变基因的信息,进一步缩小候选区域的范围,相当于在SLAF-BSA的基础上精细点,如果老师经费允许的话,群体也做重测序效果肯定是更好的,群体的SNP信息要远高于SLAF的,定位也会相应变精细点。
没有参考基因组的话,通过SLAF我们只能关联到一些标签,对于老师后期的数据利用并不是特比好,而通过转录组可以进行Uningene的组装,进而通过关联分析获得候选区域内的Uningene信息并进行功能注释,对于老师后期数据利用还是比较好的。
化学诱变或者是物理诱变引起的点突变;野生型亲本重测序+突变型子代混池测序;有参考基因组;任何发生性状分离的家系群体。
- Mol Plant丨转录组学和群体研究联合助力高粱功能基因组学研究和遗传育种2025年3月7日,中国科学院遗传与发育生物学研究所谢旗研究员团队联合中国农业大学于菲菲教授团队在Molecu […]阅读更多







 京公网安备 11011302003368号
京公网安备 11011302003368号