真核无参转录组
产品介绍
转录组测序的研究对象为特定细胞在某一功能状态下所能转录出来的所有mRNA。针对无参考基因组的物种,将小片段拼接出unigene,构建参考序列,以便后续分析,是研究无参物种分子机制与调控网络的有效手段。目前已广泛应用于基础研究、临床诊断、药物研发和分子育种等领域。
结果展示
数据质控
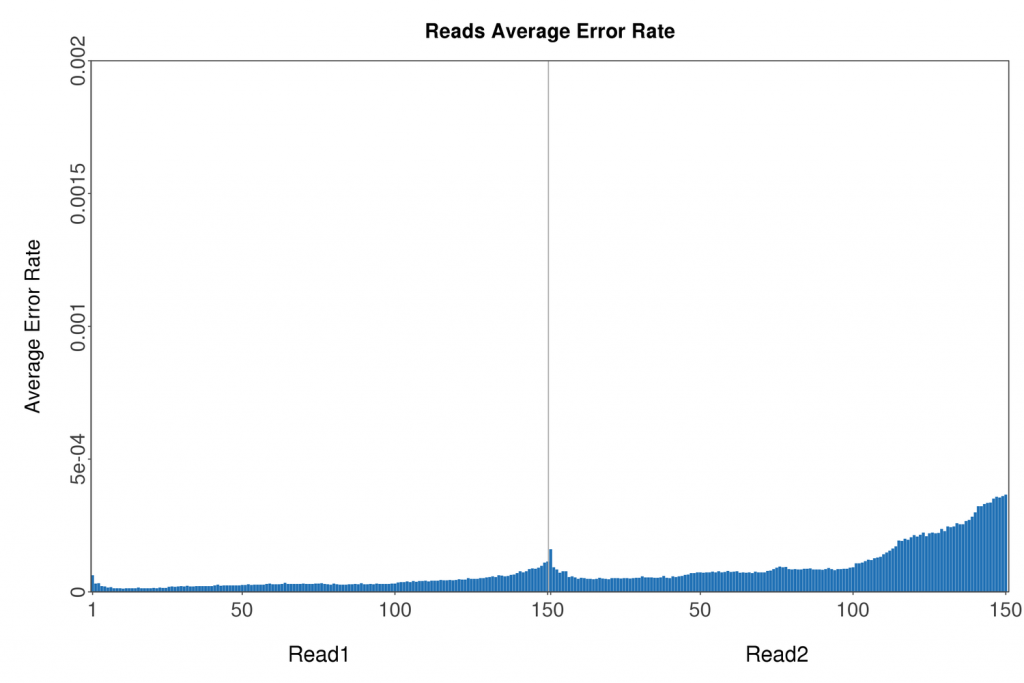
为确保Reads有足够高的质量,将下机原始测序数据(raw reads)去掉含有带接头的、低质量的reads,得到clean reads,保证后续分析的准确性。测序因受测序仪本身、测序试剂、样品等因素影响,存在一定的错误率。碱基测序错误率分布图可以反映测序数据的质量。
测序数据组装
过滤得到的高质量clean reads需通过Trinity软件进行组装得到转录本序列。转录本测序深度除了受测序数据量等影响,还与该转录本的表达丰度有关。为了使各样品中表达丰度较低的转录本组装得更完整,对于同物种的测序样品推荐合并组装可以间接增加测序深度,从而使转录结果更完整,同时也有利于后续的数据分析;而对于不同物种的样品,由于基因组间存在差异,推荐采用分别组装或分开分析。
基因表达水平分析
利用转录组数据检测基因表达具有较高的灵敏度。通过FPKM密度图和箱线图不仅可以反映单个样品基因表达水平分布和离散程度,还可以直观的比较不同样品的整体基因表达水平差异。

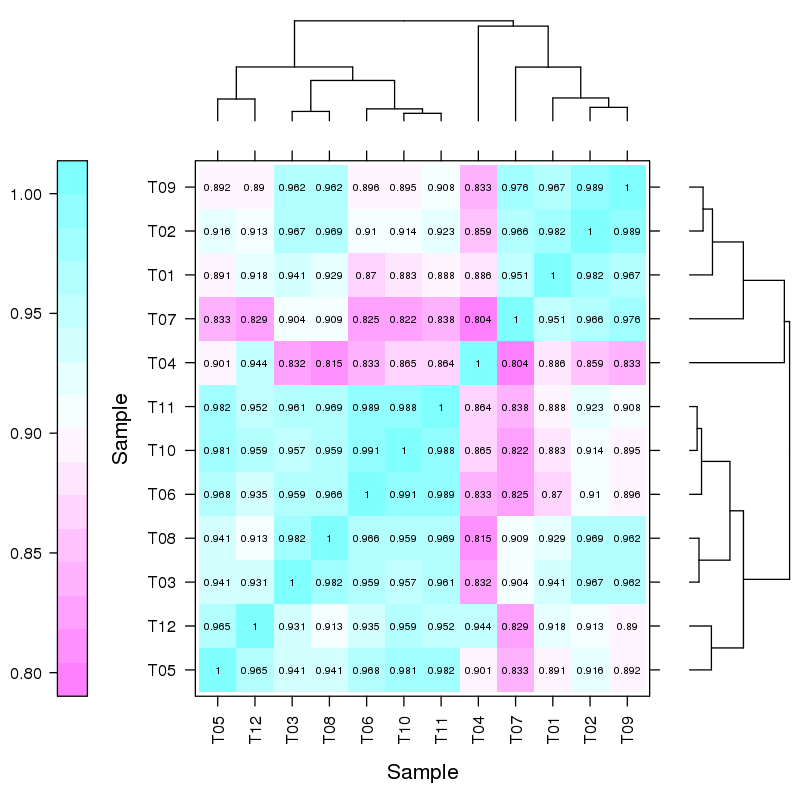
重复相关性评估
生物学重复的相关性不仅可以检验生物学实验操作的可重复性,还可以评估差异表达基因的可靠性和辅助异常样品的筛查。
差异表达基因分析
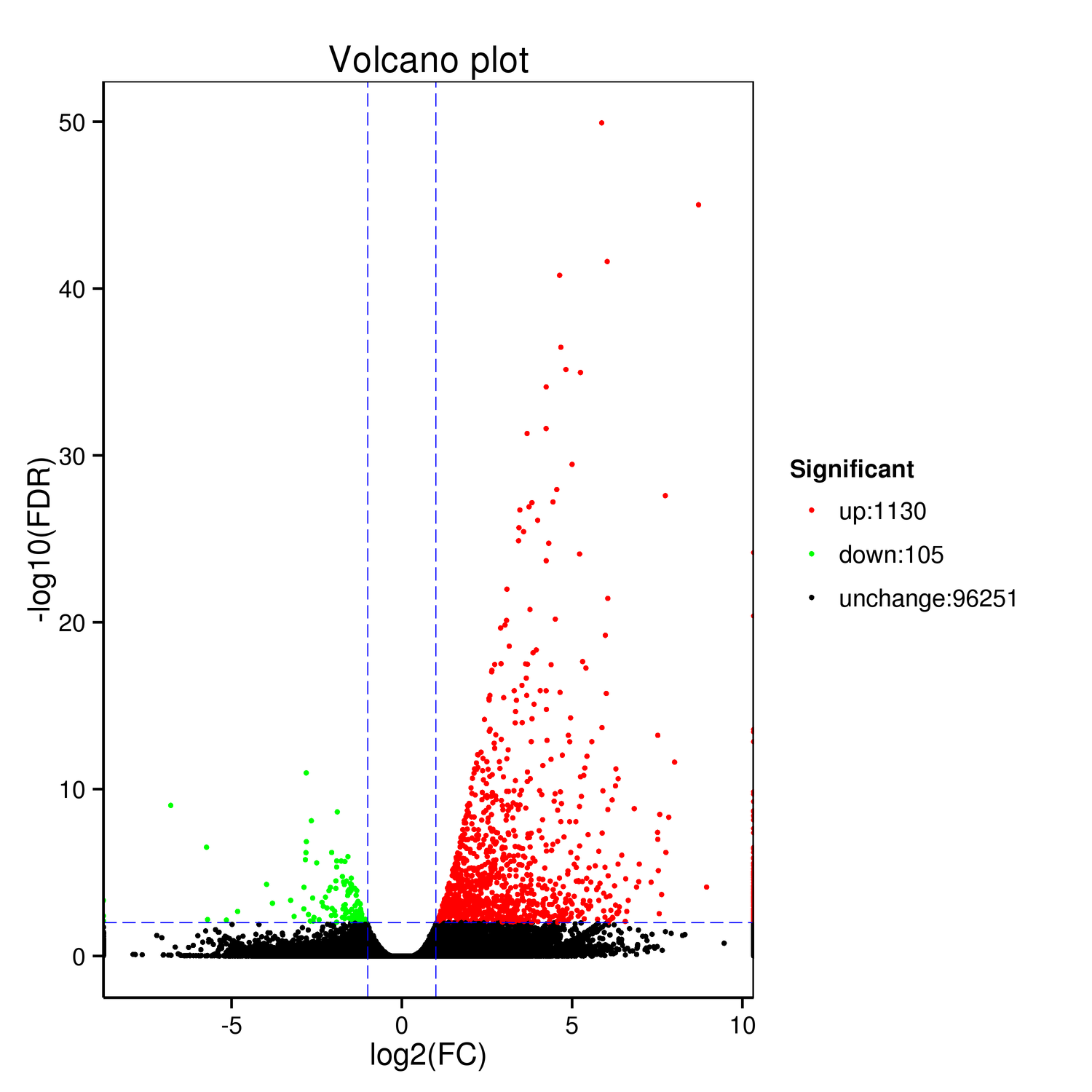
差异表达基因以火山图、MA图、韦恩图、聚类热图、蛋白互作图等形式呈现,通过火山图(Volcano Plot)可以快速地查看基因在两个(组)样品中表达水平的差异,以及差异的统计学显著性。对于有生物学重复的样本,我们采用DEseq进行样品组间的差异表达分析,获得两个生物学条件之间的差异表达基因集;对于没有生物学重复的样本,使用EBseq进行差异分析。筛选差异基因标准一般为:Fold Change≥2,FDR<0.01。
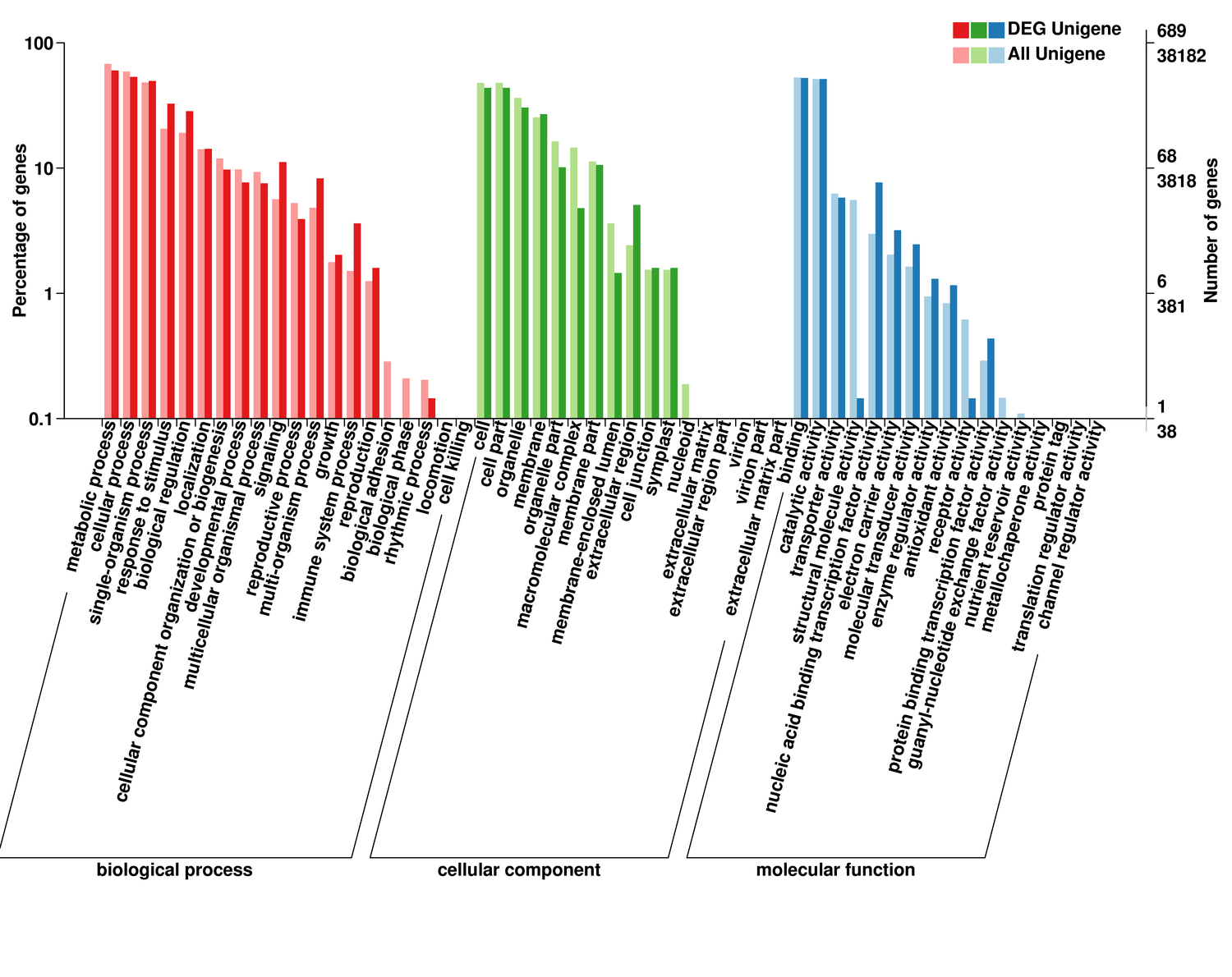
差异表达基因注释及富集分析
差异表达基因GO注释分类统计图,直观的反映出在生物过程(biological process)、细胞组分(cellular component)
和分子功能(molecular function),所有基因和差异基因注释GO term的个数分布。可深入挖掘差异基因的功能及所在的信号通路,筛选关注差异基因注释情况。
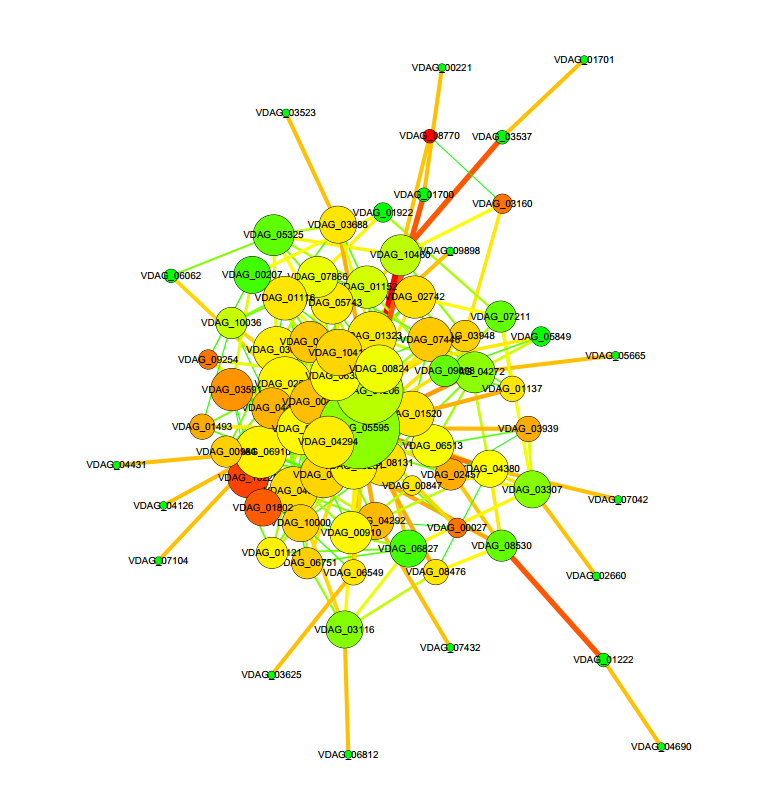
差异表达基因蛋白互作网络
STRING收录多个物种预测的和实验验证的蛋白质-蛋白质互作的数据库,包括直接的物理互作和间接的功能相关。结合差异表达分析结果和数据库收录的互作关系对,构建差异表达基因互作网络。
答:可从所有基因,差异基因及SNP三个方面进行数据挖掘。所有基因可通过功能注释信息,基因ID,基因名称,序列信息几个方面进行挖掘,同时还可以做表达基因集维恩图,WGCNA等分析。差异基因则可通过维恩图分析不同处理批次几个差异组合共同的差异基因;通常表达量变化趋势一致的基因,可能会有相似的功能,故可通过基因共表达趋势分析来进行差异基因的深入挖掘。SNP则可通过PCA分析,系统进化树,样品间差异SNP筛选及目标区域SNP查询等进行挖掘。以上这些分析均可在我公司云平台免费完成。
答: K number Count指相关的酶的数目,比如8(6)代表8个基因注释到这个通路,涉及到这个通路的6个酶,某两个基因(或多个)涉及到同一个酶。
答:Go富集我们使用的是Blast2GO R包;KEGG是我们根据fisher检验算法自己编写的程序。
KS<0.05,这个值和p-value的意义相同,是TopGO软件包中的一个检验方法。
Q-value<0.01,这个值是对p-value值的一个校正,和FDR概念相似,是fisher检验中的一个检验方法。
测序文章一般不用这两个指标,涉及到算法的文献中才有。
答:(1)用实验的方法: 针对5’端和3’端的序列来设计引物,通过PCR实验进行延长和扩增.
(2)生信办法: 将该基因与它的近源物种做同源,如果能找到同源基因,则将该区域的所有read比对到同源基因上,进而来确定中间部分的序列。





 京公网安备 11011302003368号
京公网安备 11011302003368号